NACHWEIS UND UNTERSCHEIDUNG VON LEBENDEN UND TOTEN BAKTERIEN MIT DER PCR AM BEISPIEL VON CAMPYLOBACTER SPP*
Defektion and Differentiation of viable and nonviable bacteria by PCR-for Example Campylobacter
Aus der Abt. II- Veterinäemedizin¹ (Abteilungsleiter: Dr. N. Langfeld) am Zentralen Institut des Sanitätsdienstes der Bundeswehr Kiel (Institutsleiter. Oberstveterinär Dr. H. - H. Pott), und dem Institut für Lebensmittelqualität und- sicherheit², Stiftung Tierärztliche Hochschule Hannover (Direktor: Oberstveterinär d. R. Prof. G. Klein)
Diana Seinige², Corinna Kehrenberg², Carsten Krischek², Alfred Binder¹ und Günther Klein²
Campylobacter spp. sind in Deutschland die wichtigsten bakteriellen Lebensmittelinfektionserreger. Eine Übertragung des Erregers auf den Menschen erfolgt in der Regel über Lebensmittel tierischer Herkunft oder Oberflächenwasser.
Für den Keimnachweis und die Keimzahlbestimmung aus Lebensmitteln steht ein amtliches Verfahren nach §64 LFGB zur Verfügung. Die Kultivierung ist aber sehr zeitaufwendig und benötigt etwa 6 bis 7 Tage. PCR-basierte Methoden können hingegen die Untersuchungszeit stark verkürzen, weisen aber auch DNA toter Zellen nach. Daher müssen mittels PCR nachgewiesene Campylo - bacter spp. kulturell bestätigt werden. Ziel des Forschungsvorhabens ist es, eine standardisierte quantitative Nachweismethode von Campylobacter spp. auf Geflügelfleisch und in Wasserproben zu entwickeln. Die Methode ist eine Kombination aus einer Real time-PCR und einer Bakterienbehandlung mit dem Differenzierungsmittel Ethidiummonoazid (EMA) oder Propidiummonoazid (PMA). Ziel dieser Behandlung ist es, möglichst nur lebende Zellen nachzuweisen. Verschiebungen der Detektionskurven in der PCR erfordern aber eine Anpassung über Standardkurven. Die Ergebnisse zeigen, dass Nachweis und Quantifizierung von lebenden Campylobacter spp. mittels Real time-PCR möglich sind.
Summary
Campylobacter has emerged as the most common cause of bacterial food borne disease in Germany. Transmission of Campylobacter normally occurs via contaminated food of animal origin or surface water. According to the “Amtliche Sammlung von Untersuchungsverfahren nach § 64 LFGB” a direct cultivation method has been approved for the detection and quantification of the pathogen. Nevertheless, the cultivation is time-consuming and takes up to 6 to 7 days. PCR-based methods give fast results, but are not able to differentiate between viable and nonviable cells. Therefore, it was aim of the present study to develop a rapid method for detection of Campylobacter. It is designed as a real time-PCR combined with the use of the intercalating dyes ethidium monoazid (EMA) and propidium monoazid (PMA), respect ively. This allows only the detection and quantification of viable cells. Using intercalating dyes, a shift of the sample detection curves during real time-PCR is observed, but can be compensated by type curves. This allows a calculation of viable Campylobacter cell counts by real time-PCR
1. Einführung
Campylobacter spp. gehören weltweit zu den bedeutendsten bakteriellen Lebensmittelinfektionserregern. Während sie bei einigen Haustieren, insbesondere beim Geflügel, zu den natürlichen Darmbewohnern zählen und dort nur in Ausnahmefällen klinische Erscheinungen hervorrufen, können sie für den Menschen zu einer ernsthaften Gesundheitsgefahr werden. In der Regel wird der Erreger über Lebensmittel aufgenommen und führt dann zu teils schweren Magen-Darm-Erkrankungen mit wässrigem bis blutigem Durchfall. In einigen Fällen treten dabei auch schwerwiegende Komplikationen wie Gelenksentzündungen oder das Guillain- Barré-Syndrom auf. Unter dieser Bezeichnung wird eine degenerativ-entzündliche Erkrankung des peripheren Nervensystems verstanden, die schwerwiegende Lähmungen des Patienten bis hin zum Tode verursachen kann.
Während lange Zeit Salmonellen als wichtigste bakterielle Lebensmittelinfektionserreger galten, hat sich dieses Bild innerhalb der letzten Jahre gewandelt. Inzwischen wurde die Salmonellose durch die Campylobakteriose von Platz Eins verdrängt. Letztere rief nach einer Statistik des Robert Koch-Institutes (RKI) im Jahre 2011 [1] 71 307 Erkrankungsfälle hervor. Bei den Salmonellen waren es im Jahre 2011 lediglich noch 24 512 gemeldete Erkrankungsfälle. Somit wird die Campylobakteriose nach den Norovirusinfektionen als zweithäufigste Durchfallerkrankung an das RKI übermittelt und ist gleichzeitig die wichtigste lebensmittelbedingte Erkrankung.
Die Übertragung von Campylobacter auf den Menschen erfolgt in der Regel über kontaminierte Lebensmittel tier - ischer Herkunft, vor allem über nicht ausreichend gegartes Geflügelfleisch und -produkte, die bei unzureichender Küchenhygiene auch zu Kreuzkontaminationen anderer Produkte führen können. Auch Rohmilch und Oberflächenwasser sind bei der Übertragung von Bedeutung. Die Mehrzahl der Erkrankungen tritt bei Kindern unter 5 Jahren und jungen Erwachsenen im Alter von 20-24 Jahren auf, wobei Männer in allen Altersgruppen etwas häufiger betroffen sind als Frauen [2]. Ein großer Teil der Soldaten gehört zu dieser Altersgruppe und ist damit potenziell gefährdet. Häufungen von Erkrankungen wurden ebenfalls verzeichnet, wobei die Belieferung durch Großküchen und die Teilnahme an der Gemeinschaftsverpflegung zum Teil eine ursächliche Rolle spielten.
1.1. Campylobacter - anspruchsvoll aber dennoch ein Überlebenskünstler
Campylobacter kann, im Vergleich zu anderen Lebensmittelinfektionserregern, als ein eher ungewöhnlicher Keim angesehen werden. Das Überleben dieses Erregers in der Umwelt und außerhalb seines natürlichen Lebensraumes, dem Darmtrakt des besiedelten Tieres, erscheint zunächst eher unwahrscheinlich. Zum Wachstum des Keims ist eine mikroaerobe Atmosphäre mit nur 5 % Sauerstoff und erhöhtem Kohlenstoffdioxidanteil von 10 % nötig. Durch ihre Empfindlichkeit gegenüber Sauerstoff sind für die Anzucht der Erreger anspruchsvolle Nährmedien zu verwenden, die Schutzstoffe gegen Sauerstoffradikale enthalten. Weiterhin sind Campylobacter spp. empfindlich gegenüber Austrocknung, Säuren, Hitze und hohen Salzkonzentrationen. Ihre Stoffwechselaktivität bleibt jedoch bei Temperaturen um 4°C erhalten, sodass die Bakterien eine Lagerung bei Kühlschranktemperaturen zum Beispiel besser vertragen als bei Raumtemperatur. Auch die Tiefkühlung können Campylobacter zum Teil überleben. Sie sind zudem in der Lage unter anderem bei Einwirkung von äußeren Stressfaktoren ein spe - zielles Stadium einzunehmen, das als „viable-but-non-culturable“ (VBNC) bezeichnet wird. In dieser Form sind diese Bakterien zwar nicht mehr kulturell anzuzüchten und haben eine verminderte Stoffwechselaktivität, aber bei Änderung der äußeren Überlebensbedingungen können sie wieder in ein vermehrungsfähiges Stadium übergehen. Die Bakterien bleiben für den Menschen potenziell infektiös. Somit hat Campylobacter Wege und Nischen gefunden, in der Umwelt zu überleben, obwohl eigentlich recht hohe Anforderungen an die Wachstumsbedingungen gestellt werden.
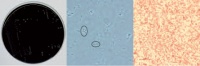
Abb 1: Morphologie von thermophilen Campylobacter-Spezies: Kolonien auf kohlehaltigem Nährboden (links); mikroskopisches Präparat (Phasenkontrast): typische Spirillenform (eingekreist) (Mitte); Gram-Färbung: gramnegative schlanke Stäbchen (rechts).
1.2. Humanpathogene Arten und ihr Vorkommen
Die wichtigsten humanpathogenen Campylobacter-Spezies sind Campy - lobacter (C.) jejuni, C. coli und C. lari. Es handelt sich dabei um gramnegative, schraubig-gedrehte Stäbchenbakterien der Gattung Campylobacter (Abb 1), die eine Breite von 0,2-0,9 µm und eine Länge zwischen 0,5 und 5,0 µm aufweisen. Diese Keime vermehren sich bei einem Temperaturoptimum von 42°C, sodass sie zu den thermophilen Arten gezählt werden. Diese thermophilen Spezies wurden im Jahr 2010 bei 99 % der Campylobacter-Erkrankungen des Menschen nachgewiesen. Dabei wurde überwiegend C. jejuni als ursächlicher Erreger identifiziert [1].
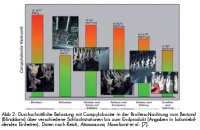
Vögel und vor allem Wirtschaftsgeflügel wie Hühner sind ideale Wirte für Campylobacter, da in deren Darmtrakt optimale Bedingungen für das Überleben herrschen. Im Blinddarminhalt von Masthähnchen werden häufig große Mengen der Erreger nachgewiesen, bis zu über 106 Bakterien/g Darminhalt können dabei ermittelt werden. Typischerweise erkranken die Tiere selbst nicht, sondern sind lediglich symptomlose Träger des Keimes. Über Schadnager, Insekten oder Personal ist ein Eintrag des Erregers in die Mastherde möglich. Eine Kolonisation des gesamten Bestandes kann dann in kürzester Zeit erfolgen. Dadurch ist oft der gesamte Bestand innerhalb von einer Woche Campylobacter-positiv. Für die Kontamination des Lebensmittels Geflügelfleisch ist ein kritischer Punkt in der Schlachtung zu sehen. Vor allem während der Prozessschritte des Brühens, des Entfederns und des Ausweidens können Kontaminationen der Tierkörperoberfläche durch von mit Campylobacter besiedeltem Blinddarminhalt entstehen. Weiterhin sind Kreuzkontaminationen durch Spülwasser und Gerätschaften möglich (Abb 2).
Um Geflügelfleisch als ein „sicheres“ Lebensmittel genießen zu können, ist deshalb dringend zu raten, durch vollständiges Erhitzen oder Garkochen die Erreger sicher abzutöten. Wichtig ist es auch, das Fleisch getrennt von anderen Lebensmitteln aufzubewahren und zuzubereiten, damit der Krankheitserreger nicht von einem Lebensmittel auf ein anderes verschleppt wird. Schneidebretter und -messer, mit denen rohes Fleisch bearbeitet wurde, sollten zum Beispiel danach nicht mehr zur Zubereitung anderer Speisen verwendet werden.
2. Methoden und Ergebnisse
Zum Nachweis von Campylobacter spp. in Lebensmitteln tierischen Ursprungs ist in der amtlichen Sammlung von Untersuchungsverfahren nach § 64 des Lebensmittelund Futtermittelgesetzbuches (LFGB) sowie des ISO-Standards ein mikrobiologisches Verfahren für den Keimnachweis und die Keimzahlbestimmung verfügbar. Der Nachteil dieses Verfahrens ist die lange Zeitdauer, bis ein Probenergebnis und die endgültige Bestätigung vorliegen (Dauer: 6-7 Tage). Durch diesen Zeitverzug ist es kaum möglich, schnell und direkt in den Produktionsund Vermarktungsprozess des Lebensmittels eingreifen zu können.
Der Nachweis des Erregers auf DNA-Ebene, wie zum Beispiel mit der Polymerase-Kettenreaktion (PCR), ermöglicht ein schnelleres Probenergebnis innerhalb weniger Stunden. Jedoch hat diese Methode den Nachteil, dass Campylobacter-DNA nachgewiesen wird, egal ob es sich dabei um einen lebenden und somit auch für den Menschen infektiösen Keim handelt, oder ob das Bakterium schon abgestorben ist und dadurch nicht mehr zu einer Erkrankung führen kann [9]. Es wird also nur die gesamte DNA aller Keime von lebenden und toten Campylobacter ermittelt. Eine Aussage über den Vitalitätsstatus der Zellen kann mit der PCR nicht getroffen werden [3, 6].
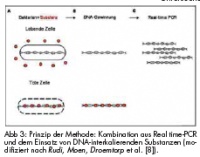
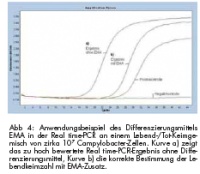
Um dieses Problem zu lösen, war es Ziel des Vertragsforschungsvorhabens* „Quantifizierung und Unterscheidung von lebenden und toten Lebensmittelinfektionserregern mittels PCR im Einsatz“, eine Schnellmethode für den Nachweis von Campylobacter zu entwickeln und im Schwerpunktinstitut Veterinärmedizin der Bundeswehr in Kiel zu etablieren. Der Ansatz war hierfür die Kombination der Real time-PCR-Methode mit dem Einsatz von so genannten DNA-interkalierenden Substanzen. Solche Substanzen können, wenn sie den Bakterien zugegeben werden, nach Belichtung irreversibel an die DNA binden. Hierbei wurden unterschiedliche DNA-interkalierende Substanzen wie Ethidiummonoazid (EMA) und Propidiummonoazid (PMA) verwendet, um zu ermitteln, welche sich für die genannte Fragestellung als geeignet erweisen [4]. Die Bindung an die DNA ist nur möglich bei membrandurchlässigen Zellen mit defekter oder fehlender Zellwand oder bei freiliegender DNA, also bei nicht mehr lebensfähigen Zellen. Bei lebenden und membranintakten Zellen hingegen können sie die Zellmembran nicht passieren und somit bleibt deren DNA ungebunden und frei von diesen inhibierenden Stoffen (Abb 3 A). Die Inkubation mit den Stoffen erfolgt für 15 min im Dunkeln. Vor der DNA-Aufreinigung wird die interkalierende Substanz durch Bestrahlung mit einer energiereichen Halogenlampe fest an die bakterielle DNA gebunden und der überschüssige Teil inaktiviert. Wird nun in einem nachfolgenden Schritt die DNA dieser Bakterien isoliert, erhält man zum einen gebundene DNA, die von toten, membrangeschädigten Zellen stammt und ungebundene DNA, die von lebenden Zellen isoliert wurde (Abb 3 B). Im nächsten Schritt erfolgt eine Real time-PCR. Hierbei ist es nicht möglich, die an interkalierende Stoffe gebundene DNA zu amplifizieren, so dass nur DNA aus lebenden Zellen, also ungebundene DNA, nachgewiesen werden kann (Abb 3 C). Mit dieser 2005 von Rudi, Moen, Droemtorp et al. [8] publizierten Methode lassen sich durch die Vorbehandlung der Zellen mit den interkalierenden Substanzen lebende, infektiöse Bakterien von toten Zellen unterscheiden und deren Keimzahl mit Hilfe der Real time-PCR in „Echtzeit“ sichtbar machen (Abb 4) [5].
3. Diskussion
Die einwandfreie Beschaffenheit der Lebensmittel ist wichtiger Bestandteil des gesundheitlichen Verbraucherschutzes und hat damit auch in der Bundeswehr entscheidende Bedeutung für die Einsatz- und Leistungsfähigkeit von Soldaten. Hervorzuheben ist in dieser Hinsicht der mikrobiologische Status der Lebensmittel. Neben Verderbniserregern, die oft bereits zu starken, sensorisch erkennbaren Veränderungen führen, spielen pathogene Keime eine entscheidende Rolle. Zu den medizinisch bedeutsamen Erregern gehören Campylobacter spp., die in Deutschland Salmonellen als häufigsten Lebensmittelinfektionserreger verdrängt haben. In der Gemeinschaftsverpflegung sind oft viele Personen betroffen. Eine große Rolle bei der Verhütung derartiger Vorfälle spielt daher eine gute Lebensmittelhygiene, besonders in den Einsatzländern mit hohen, für Mikroorganismen günstigen Temperaturen und teilweise hoher Luftfeuchte.
Das Forschungsprojekt hatte zum Ziel, die Methode der lebend-tot-Unterscheidung von Bakterien für Campylobacter zu etablieren und an verschiedenen Lebensmitteln wie zum Beispiel Geflügelfleisch und Wasserproben zu testen. Mit Hilfe dieser Studien war es möglich, ein Verfahren zur Bestimmung der Lebendkeimzahl von Campylobacter auf Geflügelfleisch zu entwickeln. Dieser Erreger wurde aufgrund der medizinischen Bedeutung und aufwändigen sowie zeitintensiven Kultivierung für die Untersuchungen ausgewählt. Das Prinzip könnte auch bei anderen Bakterien angewendet werden und eine universelle Methode zur Differenzierung lebender und toter Bakterien darstellen. Durch die Etablierung dieser Methode kann neben der Material- und Personalersparnis ein sehr schnelles Probenergebnis im Vergleich zum herkömmlichen kulturellen Verfahren erzielt werden. Dadurch sind ein zeitnaher und direkter Eingriff in die Lebensmittelsicherheit möglich und somit auch ein stärkerer Verbraucherschutz des Konsumenten gewährleistet.
4. Schlussfolgerungen
Mit der Einführung von umfangreichen kulturellen und in jüngerer Vergangenheit auch molekularbiologischen Methoden kann im veterinärmedizinischen Labor (VetMedLab) die Lebensmittelsicherheit in der Bundeswehr mit hohem Standard überprüft werden. Mit Einführung von PCR-Nachweisen im VetMedLab im Einsatz konnte auch hier das Spektrum der diagnostischen Möglichkeiten erheblich erweitert werden. In dem Forschungsvorhaben wurden grundlegende Arbeiten zur Etablierung eines PCRNachweises zur Unterscheidung von toten (nicht infektiösen) und lebenden (infektiösen) Bakterien durchgeführt. Das Verfahren zur Unterscheidung von lebenden und toten Bakterien ist ein erheblicher Fortschritt in der Diagnostik und ließe sich auch für den Nachweis anderer Bakterien anwenden, unter anderem für spezielle Fragestellungen im Bereich der klinischen Mikrobiologie. Hiermit handelt es sich um ein universelles Verfahren der Diagnostik und in diesem Fall speziell der Lebensmittelhygiene. Die Sicherheit der Produkte kann von der Herstellung bis zum Verzehr zeitnah überprüft werden.
Literatur:
- Anonymus: Infektionsepidemiologisches Jahrbuch meldepflichtiger Krankheiten für 2011. Robert Koch-Institut: 44; 165.
- Anonymus: Infektionsepidemiologisches Jahrbuch meldepflichtiger Krankheiten für 2010. Robert Koch-Institut. Epid Bull 2011; 15: 130-133.
- Cenciarini-Borde C, Courtois S, La Scola B: Nucleic acids as viability markers for bacteria detection using molecular tools. Future Microbiol 2009; 4: 45-64.
- Hein I, Schneeweiss W, Stanek C, Wagner M: Ethidium monoazide and propidium monoazide for elimination of unspecific DNA background in quantitative universal real- time PCR. J Microbiol Methods 2007; 71: 336-339.
- He Y, Chen CY: Quantitative analysis of viable, stressed and dead cells of Campylobacter jejuni strain 81-176. Food Microbiol 2010; 27: 439-446.
- Nocker A, Camper A: Novel approaches toward preferential detection of viable cells using nucleic acid amplification techniques. FEMS Microbiol Lett 2009; 291: 137-142.
- Reich F, Atanassova V, Haunhorst E, Klein G: The effects of Campylobacter numbers in caeca on the contamination of broiler carcasses with Campylobacter. International Journal of Food Microbiology 2008; 127: 116-120.
- Rudi K, Moen B, Drømtorp SM, Holck AL: Use of ethidium monoazide and PCR in combination for quantification of viable and dead cells in complex samples. Appl Environm Microbiol 2005; 71: 1018-1024.
- Wolffs P, Norling B, Rådström P: Risk assessment of false-positive quantitative real- time PCR results in food, due to detection of DNA originating from dead cells. J. Microbiol Methods 2005; 60: 315-323.
* Entstanden im Rahmen eines Forschungsvorhabens (Projektnummer M-SABX-9A006, Tierärztliche Hochschule Hannover, Institut für Lebensmittelqualität und -sicherheit) für den Sanitätsdienst der Bundeswehr.
Datum: 21.09.2012
Quelle: Wehrmedizinische Monatsschrift 2012/8-9