MOLEKULARBIOLOGISCHE RISIKOFAKTOREN EINER METASTASIERUNG BEIM SEMINOMATÖSEN KEIMZELLTUMOR DES HODENS
Molecular markers to predict metastatic status in testicular Seminoma
Aus der urologischen Abteilung (Leiter: Oberstarzt Dr. Walter Wagner) des Bundeswehrkrankenhauses Hamburg (Chefarzt: Generalarzt Dr. Joachim Hoitz) und der Arbeitsgruppe „Genomics“ (Leiter: Oberstarzt Prof. Dr. Michael Abend) des Instituts für Radiobiologe der Bundeswehr (Leiter: Oberstarzt PD Dr. Matthias Port)
Christian Ruf
WMM, 58. Jahrgang (Ausgabe 10-11/2014; S. 350-356)
Zusammenfassung
Einführung: Der Keimzelltumor des Hodens ist der häufigste Tumor des jungen Mannes und der häufigste Tumor bei Soldaten, wobei das Seminom im nicht metastasierten klinischen Stadium I (cSI) am häufigsten ist.
Zusammenfassung
Einführung: Der Keimzelltumor des Hodens ist der häufigste Tumor des jungen Mannes und der häufigste Tumor bei Soldaten, wobei das Seminom im nicht metastasierten klinischen Stadium I (cSI) am häufigsten ist. Bei 20 % der Patienten mit Seminom im cSI findet sich bei der Nachsorge eine okkulte Metastasierung, deren Risiko bisher anhand klinischer Parameter mit einer Konkordanz von bis zu 65 % abgeschätzt werden kann. Molekularbiologische Marker sollen eine bessere Vorhersage und damit eine individuellere Therapie ermöglichen.
Material und Methoden: Bei insgesamt 172 Patienten mit einem reinen testikulären Seminom (101 metastasierte und 71 nicht metastasierte) wurden Gewebe und peripheres Vollblut intraoperativ entnommen und in RNA stabilisierender Lösung gelagert. Nach der RNA Isolation erfolgte das Whole Genome Screening auf den Plattformen AB1700 Whole Genome Mikroarray (Life technologies) und Agilent Mikroarray (Agilent), sowie das Next Generation Sequencing (NGS) mittels SOLiD Platform (Life technologies). Für die quantitative Realtime Polymerase Chain Reaktion (qRTPCR) wurden die LDAs der Firma Life Technologies (Taq Chemie) genutzt. Die Durchführung und Auswertung der RNA Isolation und der Expressionsanalysen erfolgten nach den Standard Operating Procedures (SOPs) des Instituts für Radiobiologie der Bundeswehr.
Ergebnisse: Von 40 000 untersuchten Transkripten zeigten sich nach Normalisierung ca. 16 000 Transkripte exprimiert und 4 091 Transkripte differentiell exprimiert. 95 ausgewählte Gene wurden mittels qRT-PCR an einem unabhängigen Kollektiv quantitativ bestätigt. Die Konkordanz betrug bei der Expression zweier Gene (DRD1 und FAM72F1) 88 %. Bei der miRNA Analyse im Tumorgewebe und im peripheren Vollblut waren jeweils 137 small RNAs differentiell exprimiert, von denen jeweils 35, 32 und 38 small RNAs identifiziert werden konnten, die eine signifikante Diskriminierung zwischen lymphogen/okkult metastasiert bzw. beider metastasierter Subtypen von den nicht metastasierten Seminomen ermöglichten. Mittels Support Vector Machine1 war unter der Berücksichtigung zweier Gene eine vollständige Diskriminierung möglich.
Schlussfolgerungen: Die Genexpression zweier miRNAs im Tumorgewebe oder Vollblut ermöglicht eine vollständige Diskriminierung metastasierter und nicht-metastasierter Seminome. Die Bestätigung der Ergebnisse an einem größeren unabhängigen Kollektiv ist geplant. Bei Bestätigung der Ergebnisse könnte die Genexpressionsbestimmung eine individuelle Entscheidung für oder gegen eine adjuvante Therapie ermöglichen.
Schlüsselwörter: Seminom, Metastasierung, miRNA, mRNA, molekulare Marker
Summary
Introduction: The germ cell tumor is the most frequently occurring neoplasm of the young man and also of soldiers, whereby the seminoma in the non-metastasised clinical stadium I (cSI) appears most often. At follow up, 20 % of patients with seminoma in cSI show occult metastasis, which, under clinical parameters, with a an estimated risk rate of up to 65 %. Molecular biological marker should allow a better prognosis and thus a more individual therapy.
Material and Methods: From a total of 172 patients with a straight testicular seminoma (101 metastasized and 72 non-metastasized) tissue and whole blood samples were extracted intraoperatively and placed in RNA-stabilized solution. After isolating the RNA Whole Genome Screening with the platforms AB1700 Whole Genome Mikroarray (Life technologies) and Agilent Mikroarray (Agilent) was performed as well as Next Generation Sequencing (NGS) by SOLiD Platform (Life technologies).
Results: Of 40 000 transcripts explored, after normalization, 16 000 appeared expressed and 4 091 differentially expressed. Within an idependent collective, 95 selected genes could be confirmed quantitatively through qRT-PCR. The concordance for the expression of two genes (DRD1 and FAM72F1) was 88 %. From the miRNA analysis of tumor tissue and peripheral whole blood 137 small RNAs were differentially expressed; of thoses 35, 32 and 38 small RNAs could be identified respectively, which allowed a significant discrimination between lymphogenic/occult metastasis or both subtypes of metastasis and those seminomas without metastasis.
Under the condition of two genes, a complete discrimination could be achieved using Support Vector Machine.
Conclusion: The gen expression of two miRNAs in human tumor tissue or whole blood allows for complete differentiation of metastasizing and non-metastasizing seminomas.
Confirmation of results in a larger independent collective has been planned. If the results can be confirmed the identification of gene expression could enable an individual decision for or against an adjuvant therapy.
Keywords: Seminoma, metastatic status, miRNA, mRNA, molecular marker
Einleitung
Der Keimzelltumor des Hodens ist der häufigste Tumor des jungen Mannes und damit der häufigste Tumor bei Soldaten. Jährlich werden etwa 4.000 Neuerkrankungen in Deutschland registriert. Die Bundeswehrkrankenhäuser sind Zentren für die Behandlung des Hodentumors, teilweise zertifizierte Zweitmeinungszentren der Deutschen Krebsgesellschaft; mehr als 10 % aller Hodentumorpatienten werden in Deutschland in einem Bundeswehrkrankenhaus behandelt. Die Aussetzung der Wehrpflicht hatte dabei bisher keinen Einfluss auf die Behandlungszahlen. Fragestellungen zur Diagnostik und Behandlung des Hodentumors sind im klinischen Alltag in den Bundeswehrkrankenhäusern von hoher Relevanz.
Der Keimzelltumor des Hodens wird je nach histologischem Subtyp in Seminome und Nicht-Seminome eingeteilt. Heute liegt in 56 % der Fälle ein reines Seminom vor [1].
Die Heilungschancen früher Stadien, wie dem nicht metastasierten Seminom, liegen bei frühzeitiger Diagnosestellung und stadiengerechter Therapie bei bis zu 100 %.
Die meisten Tumoren fallen durch eine Vergrößerung oder Verhärtung des Hodens, die vom Patienten oder dem/der Partner( in) bemerkt wird, auf. Immer häufiger werden Tumoren auch sonografisch im Rahmen der allgemeinen Vorsorgeuntersuchung oder der urologischen Abklärung bei unerfülltem Kinderwunsch diagnostiziert. Die Diagnose wird mittels Palpation des Hodens, Ultraschall und den Tumormarkern Alphafetoprotein (AFP), Humanes Chorion Gonadotropin (ß-HCG) und Lactatdehydrogenase (LDH) gestellt.
Etwa 20 % der Seminompatienten im cSI entwickeln ohne adjuvante Therapie einen Progress der Erkrankung [2] und werden als okkult metastasiert (die vorhandene Metastasierung ist zum Zeitpunkt des Stagings noch nicht sichtbar) bezeichnet. Nicht metastasierte Patienten sind durch die Ablatio testis geheilt und benötigen keine weitere Therapie. Okkult metastasierte Patienten profitieren von einer adjuvanten Therapie, die das Risiko eines Progresses auf unter 5 % reduziert. Zur Unterscheidung zwischen okkult und nicht metastasierten Patienten wurden im Jahr 2002 von Warde et al. die Risikofaktoren Tumorgröße 4 cm und die Infiltration des Rete testis identifiziert [3]. Bei Vorliegen von einem Risikofaktor steigt das Risiko einer okkulten Metastasierung von 12 % auf 20 % und beim Vorliegen von zwei Risikofaktoren auf 30 % an. Die Validierung dieser Risikofaktoren an einem unabhängigen Kollektiv gelang der gleichen Arbeitsgruppe im Jahr 2010 nicht [4]. In einem bundeswehreigenen Kollektiv konnten wir 90 klinische, histologische, epidemiologische und laborchemische Parameter hinsichtlich Ihrer Assoziation mit dem Metastasierungsstatus untersuchen. In diese Studie mit 527 Seminompatienten wurden im Jahr 2013 die Parameter Tumorlänge, Lymph- und Blutgefäßinvasion sowie die Tumormarker ß-HCG und LDH als Risikofaktoren identifiziert. Die Diskriminierungsfähigkeit der metastasierten von den nicht-metastasierten Seminomen lag in der multivariaten Analyse trotzdem bei maximal 65 % (Konkordanz oder ROC area) [5]. Auch andere Arbeitsgruppen kamen zu ähnlichen Ergebnissen [6]. Dieses Ergebnis zeigt einmal mehr, dass signifikante Assoziationen und die Diskriminierungsfähigkeit zwei unterschiedliche Aspekte darstellen. Zudem unterstreicht es die Dringlichkeit, diagnostisch relevantere Parameter zu finden.
Eine adjuvante Therapie mit einem Zyklus Carboplatin-Monochemotherapie senkt das Risiko eines Progresses bzw. Rezidivs im cSI von 20 - 30 % auf unter 5 %. Die Langzeittoxizitäten einer adjuvanten Chemotherapie sind neben kardiovaskulären Nebenwirkungen die Einschränkungen der Fertilität und der Nierenfunktion sowie ein erhöhtes Risiko von Zweitmalignomen [7-12]. Das Auftreten von Nebenwirkungen und Langzeittoxizitäten einer Therapie ist gerade beim Hodentumor aufgrund des jungen Patientenalters und der sehr guten Heilungschancen relevant. Mit einer adjuvanten Therapie sind 80 % der Patienten übertherapiert. Alternativ zu einer adjuvanten Chemotherapie kann eine aktive Überwachung (Active Surveillance) durchgeführt werden. Im Rahmen der Nachsorge wird dann regelmäßig eine Bildgebung mit CT Abdomen/Becken durchgeführt - eine für 80 % der Patienten überflüssige Strahlenexposition mit dem Risiko einer dadurch induzierten späteren Tumorentwicklung.
Molekularbiologische Marker wie die Expression der proteinkodierenden messenger RNAs (mRNA) oder der nicht kodierenden kurzen micro RNAs (miRNA) werden bei unterschiedlichen (urologischen) Tumoren incl. Hodentumoren untersucht. Dabei waren verschiedene miRNAs mit dem Metastasierungsstatus bei anderen Tumoren assoziiert [13].
Eine Assoziation der Expression von mRNA und miRNA mit dem Metastasierungsstatus wurde bisher bei Hodentumoren nicht untersucht. Ziel der Arbeit war es, Biomarker zu identifizieren, welche eine noch bessere Vorhersage des Metastasierungsstatus ermöglichen. Der Fokus lag dabei nicht so sehr auf sichtbaren Metastasen, die in der CT diagnostiziert werden können, sondern in der Detektion okkulter Metastasen.
Ein Biomarker, der okkulte Metastasen mit hoher Sensitivität und Spezifität vorhersagen kann, hätte eine hohe klinischer Relevanz. Anhand des Markers könnte die fast arbeitstäglich anstehende Entscheidung bei Seminomen im cSI für oder gegen eine adjuvante Therapie bzw. eine engmaschige Nachsorge getroffen werden. In der Konsequenz würden nur die Patienten behandelt, bei denen es notwendig ist. Eine unnötige Exposition gegenüber therapieassoziierten Toxizitäten und ionisierender Strahlung könnte somit vermieden werden.
Die Suche nach einem molekularen Marker wurde in mehreren Teilschritten als Einzelprojekte durchgeführt.
Material und Methoden
Patienten
In die Studie wurden ausschließlich Patienten mit einem reinen testikulären Seminom eingeschlossen. Die histologische Diagnose wurde durch einen Pathologen mit viel Erfahrung in dem Bereich der histologischen Hodentumordiagnostik gestellt. Für die unterschiedlichen Projekte wurden Proben von insgesamt 172 Patienten eingeschlossen, davon 101 metastasierte und 71 nicht metastasierte Seminome.
Die Patienten wurden in drei Gruppen eingeteilt.
1) Nicht metastasierte Seminome, klinisches Stadium I (cSI), die ohne Hinweis auf Metastasen keine adjuvante Therapie erhielten und während des Follow up von mindestens 2 Jahren kein Tumorrezidiv zeigten. 2) Okkult metastasierte Seminome, die im primären Staging bildgebend und laborchemisch (Tumormarker) keinen Hinweis auf eine Metastasierung zeigten, keine adjuvante Therapie erhielten und im Follow up einen Tumorrezidiv im Sinne einer Metastasierung zeigten. 3) Sichtbar metastasierte Seminome, cSIIa–III, teilweise auch nur lymphogen metastasierte Seminome (cSIIb-IIc), Patienten, die zum Zeitpunkt des primären Stagings schon sichtbare Metastastasen hatten.
Gewebeentnahme und Aufbewahrung Das Tumorgewebe und korrespondierendes Hodennormalgewebe wurden intraoperativ aus repräsentativen vitalen Bereichen des Tumors bzw. Hodens standardisiert entnommen und direkt in RNA stabilisierende Lösung überführt. Zusätzlich wurde bei den Patienten Vollblut aus der Cubitalvene direkt in RNA stabilisierende Blutröhrchen (PAX Gene, Fa. Becton, Dickinson and Company, BD) abgenommen. Das Material wurde bis zur Aufarbeitung bei -20°C gelagert.
RNA Isolation
Die RNA Isolation aller Proben erfolgte mittels säulenbasierten RNA Extraktionskits mRNEasy bzw. miRNEasy der Firma Qiagen bzw. der Kombination einer RNA-Fällung in Phenol- Chloroform gefolgt von einer Säulenaufreinigung zur Isolation sogenannter „small RNAs“, in denen auch die uns interessierenden microRNAs enthalten sind. Alle Laborarbeiten wurden nach DinISO 9001:2000 zertifizierten Standards der Arbeitsgruppe „Genomics“ des Instituts für Radiobiologie der Bundeswehr durchgeführt.
Whole Genome Mikroarray Für die Untersuchung mittels AB1700 Whole Genome Mikroarray wurden die gepoolten Proben in copy DNA (cDNA) umgeschrieben und dabei mit einem Chemolumineszenz RT Labeling Kit markiert. Die markierte cDNA der insgesamt 4 verschiedenen gepoolten Proben wurde auf je einem Whole Genome Mikroarray (Human-Genome-Survey-Mikroarray 2.0, Lifetechnologies, Darmstadt) (Abb. 1) hybridisiert und mittels Mikroarray Reader AB1700 (Applied Biosystems, Darmstadt) ausgelesen. Nach Normalisierung gegen die Gesamtgenexpression wurde die Expression jedes einzelnen Gens bei den metastasierten und den nicht-metastasierten Proben gegeneinander verglichen.
Ein Gen wurde als exprimiert betrachtet, wenn das Chemilumineszenzsignal mehr als fünffach gegenüber dem Hintergrundrauschen erhöht war. Ein mindestens dreifacher Unterschied des normalisierten Chemilumineszenzsignals in der metastasierten gegenüber der nicht-metastasierten Gruppe wurde als differentielle Genexpression definiert.
Für den Agilent Mikroarray wurde die Gesamt-RNA revers transkribiert, die cDNA dann in Cyanine-3 markierte cRNA konvertiert, aufgereinigt und ungepoolt auf den Agilent oligo Mikroarray hybridisiert. Die Auswertung des Genom-weiten Expressionsprofils erfolgte mittels Agilent DNA Mikroarray Scanner. Gene, die in >50 % der Proben pro Gruppe exprimiert waren und mehr als zweifache Unterschiede gegenüber der anderen Gruppe zeigten, wurden als differentiell exprimierte Gene gewertet. Die nicht adjustierten und die adjustierten p-Werte (multiple comparison, false discovery rate) wurden berechnet.
Quantitative Real Time Polymerase Kettenreaktion (qRT-PCR)
Next Generation Sequencing (NGS) Aus selektierten Biopsaten wurde die Gesamt-RNA isoliert und die angereicherten small RNAs an die SOLiD adaptors ligiert. Nach der reversen Transkription wurde die cDNA aufgereinigt und RNA-Spezies größer 60 - 80 nt wurden verworfen. Nach der in-gel Amplifikation der cDNA erfolgte die Markierung mittels SOLiD 3´Barcode primer. Die amplifizierte cDNA wurde aufgereinigt und in die Emulsions-PCR eingesetzt. Die Emulsion wurde aufgebrochen und die so genannten „di-base“ Proben in das SOLiD System zur Sequenzierung nach dem sequencing- by-ligation Prinzip eingesetzt.
Zur Darstellung, Signalberechnung, für das Clustering, das Auszählen der Reads und die Benennung wurde die SOLiD5500xl Software (LifeScope) und die CLC Genomics Workbench 5.1 (CLC bio) genutzt. Anschließend erfolgte das Trimming (Aussortieren aller Signale ohne Annotation und zu kurzer Fragmente). Small RNAs mit einem Unterschied in der Genexpression >2 und mindestens 50 Reads wurden weiter untersucht.
Bioinformatik
Die Rohdaten der einzelnen Untersuchungen mussten zunächst normalisiert werden. Als Referenz wurde die Gesamtexpression der Untersuchung oder für die qRT-PCR auch ein Haushaltsgen (18S) genutzt. Nach der Normalisierung wurde die differentielle Expression der Gene untersucht, indem die normalisierte Genexpression jedes einzelnen Gens in den beiden untersuchten Gruppen miteinander verglichen wurde. Ein Unterschied >2 wurde in der Regel als differentielle Expression gewertet. Die Daten der Screeningverfahren (Whole Genome Mikroarray und Next Generation Sequencing) wurden aufgrund des möglichen Alpha-Fehlers beim multiplen Testen nach Bonferroni korrigiert. Nachdem in den Untersuchungen kein einzelnes Gen zur Differenzierung ausreichte, wurden mehrere Variablen (mRNAs, miRNAs, klinische Risikofaktoren wie Tumorgröße) mittels multivariater Analyse untersucht. Dabei wurde die „Area under the Curve“ (AUC) einer „Receiver Operator Correlation curve“ (ROC curve) unter Berücksichtigung verschiedener Variablen berechnet. Die statistische Auswertung erfolgte mittels SAS Software.
Die Differenzierung metastasierter und nicht metastasierter Semiome anhand zweier miRNAs erfolgte mittels Support Vector Machine.
Ergebnisse und Diskussion
Die mRNA Expression ist mit dem Metastasierungsstatus assoziiert, Ergebnisse der Screeninguntersuchung [14]
Zu Beginn der Studien waren in der Literatur keine Gene beschrieben, die eine Assoziation mit dem Metastasierungsstatus bei Hodentumoren aufweisen. Daher untersuchten wir an 31 Patienten (10 metastasiert und 21 nicht metastasiert) das gesamte Genom (alle 20 000 bekannten humanen mRNA`s) mittels Whole Genome Mikroarray, einem semiquantitativen Screeningverfahren. Von 20 000 untersuchten Genen bzw. 40 000 Transkripten zeigten sich nach Normalisierung ca. 16 000 Transkripte exprimiert. Beim Screening metastasierter und nicht metastasierter Seminome fanden sich 4 091 Transkripte differentiell exprimiert. Durch die begrenzte Patientenzahl sowie das gleichzeitige Untersuchen von bis zu 20 000 Genen (multiples Testen) im Rahmen des Screening könnten Verzerrungen oder Fehler (Alphafehler) aufgetreten sein. Daher war es wichtig, die differentiell exprimierten Gene quantitativ zu untersuchen. Aus den 4 091 differentiell exprimierten Genen wurden 95 Gene ausgewählt, die mittels qRT-PCR an dem gleichen Kollektiv quantitativ bestätigt wurden. Dabei wurden vor allem die Gene ausgesucht, die (1) große Expressionsunterschiede aufwiesen, (2) bekanntermaßen mit dem Metastasierungsstatus bei anderen Tumorentitäten korrelierten oder (3) bei denen eine Beteiligung an biologischen Prozessen der Metastasierung beschrieben worden ist. Aus finanziellen und logistischen Gründen konnten nicht alle 4 091 Transkripte untersucht werden. Die Ergebnisse der qRT-PCR stimmten in 88 % der untersuchten Gene mit den Ergebnissen des Mikroarrays überein. Die Rate an falsch Positiven lag bei 1 % und bei falsch Negativen bei 11 %. Durch qRT-PCR eines von jeweils fünf Genen war eine vollständige Diskriminierung von Tumor- und Normalgewebe bzw. eine Diskriminierung der unterschiedlichen Tumorentitäten möglich.
Eine Metastasierung konnte anhand der Expression einzelner Gene nicht erkannt werden. Unter Benutzung eines logistischen Regressionsmodells, basierend auf einem Genexpressionsprofil von 85 Genen, konnte eine 88 %ige Vorhersage des Metastasierungsstatus der Seminome erfolgen. Die Ergebnisse dieses zweistufigen Experiments dokumentierten eine gute Übereinstimmung (88 %) der semiquantitativen Mikroarray Ergebnisse mit den quantitativen RT-PCR Ergebnissen. Erstmals konnte gezeigt werden, dass beim Hodentumor Gene eindeutig mit dem Metastasierungsstatus assoziiert sind. Es fand sich kein einzelnes Gen als Marker einer Metastasierung, aber mittels eines bioinformatischen Modells konnte anhand eines Sets von 85 Genen der Metastasierungsstatus eindeutig vorhergesagt werden.
Die Aussagen des bioinformatischen Modells werteten wir kritisch, denn eine Signatur aus 85 Genen, die an einem Gesamtkollektiv von 31 Patienten erhoben wurde, ist aufgrund ihrer Komplexität auf der einen Seite und der geringen Fallzahl auf der anderen Seite problematisch. Wir werteten es indes als einen ersten Hinweis darauf, dass basierend auf Transkriptionsänderungen vermutlich Aussagen zur Metastasierung am Primärtumor möglich sind. In weiteren Untersuchungen wurden die Genexpressionsdaten deshalb an einem unabhängigen Kollektiv mit robusteren Verfahren wie der logistischen Regressionsanalyse untersucht.
Zwei Gene erlauben eine Vorhersage des Metastasierungsstatus zu 88 % [15]
Die oben beschriebenen Ergebnisse wurden an einem unabhängigen Kollektiv von 52 Seminompatienten (12 metastasiert , 40 nicht-metastasiert) validiert. Zusätzlich wurden die in der Literatur beschriebenen klinischen Risikofaktoren (Tumorgröße und Infiltration des Rete testis) mit untersucht.
In der Auswertung zeigte sich die Expression von fünf Genen signifikant mit dem Metastasierungsstatus assoziiert. Anders als in der Analyse zuvor wurde eine logistische Regressionsanalyse jedes einzelnen Gens und der Kombination selektierter Gene zur Diskriminierung des Metastasierungsstatus durchgeführt. Die Expression von zwei Genen (Dopamin Rezeptor D1 [DRD1] und family with sequence similarity 71, member F2 [FAM71F2], p=0,005 und 0,024 in der Einzelanalyse und p=0,004 und 0,016 in der Kombination beider Gene) ermöglichte eine signifikante Diskriminierung des Metastasierungsstatus. Die Konkordanz stieg von 77,9 % (DRD1) und 72,3 % (FAM71F2) in der Einzelanalyse auf bis zu 87,7 % bei der Kombination beider Gene.
Bei der Untersuchung der klinischen Risikofaktoren konnte nur für die Größe des Primärtumors (als kontinuierliche Variable und als Kategorie >6 cm) eine signifikante Assoziation mit der Metastasierung gezeigt werden (p=0,039/p=0,02). Allerdings war die Konkordanz niedrig (61 %). Eine Kombination der Genexpression mit dem klinischen Risikofaktor ergab keine Verbesserung der Konkordanz.
Mit dieser Studie konnten die Ergebnisse der Screeninguntersuchung bestätigt werden: Fünf der untersuchten Gene zeigten eine Assoziation mit dem Metastasierungsstatus und eine 88 %-ige Diskriminierung war anhand der Expression von zwei Genen (DRD1 und Fam71F2) der Nutzung von klinischen Risikofaktoren mit einer Diskriminierungsfähigkeit von bis zu 65 % überlegen. Durch diese Arbeit hat sich zwar die diagnostische Vorhersage verbessert, das initiale Ziel einer sicheren Diskriminierung konnte durch die mRNA Expression jedoch nicht vollständig erreicht werden. Parallel dazu verbesserte sich die Untersuchungstechnik für small RNAs und es mehrten sich die Hinweise auf die diagnostische Wertigkeit der small RNAs, auch beim Keimzelltumor des Hodens [16-19]. Daher untersuchten wir im nächsten Schritt die miRNA Expression im Primärtumor [20] mittels Next Generation Sequencing.
Die miRNA Expression im Tumorgewebe erlaubt eine vollständige Differenzierung metastasierter und nicht metastasierter Seminome [20]
Nachdem die mRNA Expression keine vollständige Diskriminierung des Metastasierungsstatus ermöglichte, untersuchten wir an einem unabhängigen Kollektiv die small RNA Expression im Primärtumor. Wir screenten die ca. 1.000 bekannten small RNAs mittels Next Generation Sequencing (NGS), einer quantitativen Untersuchungsmethode, da nicht klar war, welche small RNAs sich zur Diskriminierung metastasierter und nicht-metastasierter Seminome eignen würde. Darüber hinaus erlaubt dieses Verfahren auch die Identifizierung bislang unbekannter und für unsere Zwecke vielleicht geeignetere small RNA-Spezies.
Der wesentliche Nutzen eines molekularen Markers besteht in der Detektion okkulter Metastasen, die also während der Diagnostik des Primärtumors nicht nachweisbar sind im Gegensatz zu den zu diesem Zeitpunkt sichtbaren (nachweisbaren) Metastasen. Ob diese sich auf transkriptionaler Ebene von den sichtbar metastasierten Seminomen unterscheiden, war unklar. Daher führten wir NGS an je 5 Seminompatienten aus den drei Gruppen, sichtbar metastasiert, okkult metastasiert und nicht-metastasiert durch. Für die metastasierten Seminome beschränkten wir uns nur auf die klinischen Stadien IIb und IIc. Diese Stadien sind lymphogen metastasiert (wie auch die okkult metastasierten) und grenzwertige Metastasierung, wie teilweise beim cSIIa, werden nicht eingeschlossen.
Durchschnittlich fanden sich 1,3x107, 1,4x107 und 1,7x107 Reads bei den lymphogen metastasierten, okkult metastasierten und nicht metastasierten Seminomen. Nach dem „Trimming“ reduzierte sich die Anzahl auf durchschnittlich 30 - 32 %, wovon 59 - 68 % annotierte Reads mit 8,6 - 11 % (3,6 - 5,7x104) annotierten small RNAs gefunden wurden. Insgesamt zeigten 137 small RNAs eine Genexpression >2 und mindestens 50 Reads. In der univariaten logistischen Regressionsanalyse konnten 35, 32 und 38 small RNAs identifiziert werden, die eine signifikante Diskriminierung zwischen lymphogen/okkult metastasiert bzw. beider metastasierter Subtypen von den nicht metastasierten Seminomen ermöglichten (Abb. 3). Eine Differenzierung zwischen okkult und sichtbar metastasierten Seminomen war nicht möglich. Schließlich nutzten wir Support Vector Machine Berechnungen zur kompletten Diskriminierung metastasierter und nicht metastasierter Stadien. Insgesamt wurden 125, 52 und 6 Kombinationen zweier small RNAs identifiziert, die eine vollständige Diskriminierung der lymphogen metastasierten, der okkult metastasierten und der insgesamt metastasierten Seminome (sowohl lymphogen als auch okkult metastasiert) von den nicht metastasierten Seminomen ermöglichte.
Somit ist anhand der Expression zweier small RNAs im Primärtumor erstmals eine vollständige Diskriminierung metastasierter von nicht metastasierten Seminomen möglich. Die Anzahl der eingeschlossenen Patienten wurde aufgrund der hohen Kosten dieser Technik auf 15 reduziert. Die Bestätigung der Ergebnisse muss in einem nächsten Schritt mittels qRT-PCR an einem größeren unabhängigen Kollektiv erfolgen.
Eine weitere wichtige Erkenntnis konnte aus dieser Arbeit gewonnen werden: Metastasierte Tumoren unterscheiden sich auf small RNA-Ebene von den nicht metastasierten, eine Unterscheidung sichtbar und okkult metastasierter Tumoren ist aber nicht möglich. Das bestätigte unsere Ergebnisse der klinischen Risikofaktoranalysen, bei der die gleichen Risikofaktoren sowohl für okkult als auch sichtbar metastasierte Tumoren identifiziert worden waren. Diese Ergebnisse unterstreichen zudem die These, dass der biologische Prozess der Metastasierung bei den okkult metastasierten Seminomen bereits eingetreten ist, die Metastasen nur aufgrund ihrer geringen Größe noch nicht nachweisbar sind.
Die Gewinnung von Tumorgewebe kann nur intraoperativ stattfinden und muss unter sterilen Bedingungen innerhalb von 10 Minuten Ischämiezeit aus repräsentativen Bereichen des vitalen Tumors erfolgen. Zusätzlich könnte die Gewebeentnahme die histopathologische Diagnostik stören. Nachdem sich mehrere Publikationen die diagnostische Wertigkeit für miRNAs im Hodentumorgewebe und im peripheren Blut nachweisen konnten [17, 21-24], war der nächste Schritt, die small RNA Expression auch im Blut durchzuführen [25].
Die miRNA Expression im Vollblut erlaubt eine vollständige Differenzierung metastasierter und nicht metastasierter Seminome [25]
Unsere vorherigen Studien haben gezeigt, dass die small RNA Expression im Tumorgewebe eine vollständige Diskriminierung des Metastasierungsstatus ermöglicht. Ein molekularer Marker im Blut, der im Rahmen einer Blutentnahme und nicht nur intraoperativ bestimmt werden kann, würde die klinische Anwendbarkeit vereinfachen. Gleichzeitig zeigten aktuelle Studien, dass die miRNAs sich als valider Hodentumormarker im Gewebe und im Blut eignen. Daher untersuchten wir die miRNA Expression bei metastasierten und nicht-metastasierten Seminomen im peripheren Vollblut. Es war nicht klar ob die gleichen miRNAs, die wir im Gewebe identifizierten konnten, sich auch zur Diskriminierung metastasierter und nicht metastasierter Seminome im Blut eignen würden. Daher führten wir ein Screening mittels Next Generation Sequencing (NGS) an dem peripheren Vollblut von 15 Seminompatienten durch, je 5 Patienten aus den drei Gruppen lymphogen metastasierter, okkult metastasierter und nicht metastasierter Seminome.
Durchschnittlich fanden sich 1,3x107, 1,2x107 und 1,2x107 Reads bei den lymphogen, okkult und nicht metastasierten Seminomen. Nach dem „Trimming“ blieben durchschnittlich 73 - 76 % übrig, wovon 80 - 84 % annotierte Reads mit 7,2 - 7,8 % (1,6 - 1,7x104) annotierten small RNAs. Insgesamt zeigten 137 small RNAs eine Genexpression >2 und mindestens 50 Reads. In der univariaten Analyse konnten 35, 32 und 38 small RNAs identifiziert werden, die eine signifikante Diskriminierung zwischen lymphogen/okkult metastasiert bzw. beider metastasierter Subtypen kombiniert von den nicht metastasierten Seminomen ermöglichten (Abb. 4). Eine Differenzierung zwischen okkult und sichtbar metastasierten Seminomen war nicht möglich. Schließlich nutzten wir Support Vector Machine zur kompletten Diskriminierung metastasierter und nicht metastasierter Stadien. Insgesamt waren 891, 668 und 87 Kombinationen mögten dieser Technik auf 15 reduziert. Die Bestätigung der Ergebnisse muss in einem nächsten Schritt mittels qRT-PCR an einem größeren unabhängigen Kollektiv erfolgen. Eine weitere wichtige Erkenntnis konnte aus dieser Arbeit gewonnen werden: Metastasierte Tumoren unterscheiden sich auf small RNA-Ebene von den nicht metastasierten, eine Unterscheidung sichtbar und okkult metastasierter Tumoren ist aber nicht möglich. Das bestätigte unsere Ergebnisse der klinischen Risikofaktoranalysen, bei der die gleichen Risikofaktoren sowohl für okkult als auch sichtbar metastasierte Tumoren identifiziert worden waren. Diese Ergebnisse unterstreichen zudem die These, dass der biologische Prozess der Metastasierung bei den okkult metastasierten Seminomen bereits eingetreten ist, die Metastasen nur aufgrund ihrer geringen Größe noch nicht nachweisbar sind. Die Gewinnung von Tumorgewebe kann nur intraoperativ stattfinden und muss unter sterilen Bedingungen innerhalb von 10 Minuten Ischämiezeit aus repräsentativen Bereichen des vitalen Tumors erfolgen. Zusätzlich könnte die Gewebeentnahme die histopathologische Diagnostik stören. Nachdem sich mehrere Publikationen die diagnostische Wertigkeit für miRNAs im Hodentumorgewebe und im peripheren Blut nachweisen konnten [17, 21-24], war der nächste Schritt, die small RNA Expression auch im Blut durchzuführen [25]. Die miRNA Expression im Vollblut erlaubt eine vollständige Differenzierung metastasierter und nicht metastasierter Seminome [25] Unsere vorherigen Studien haben gezeigt, dass die small RNA Expression im Tumorgewebe eine vollständige Diskriminierung des Metastasierungsstatus ermöglicht. Ein molekularer Marker im Blut, der im Rahmen einer Blutentnahme und nicht nur intraoperativ bestimmt werden kann, würde die klinische Anwendbarkeit vereinfachen. Gleichzeitig zeigten aktuelle Studien, dass die miRNAs sich als valider Hodentumormarker im Gewebe und im Blut eignen. Daher untersuchten wir die miRNA Expression bei metastasierten und nicht-metastasierten Seminomen im peripheren Vollblut. Es war nicht klar ob die gleichen miRNAs, die wir im Gewebe identifizierten konnten, sich auch zur Diskriminierung metastasierter und nicht metastasierter Seminome im Blut eignen würden. Daher führten wir ein Screening mittels Next Generation Sequencing (NGS) an dem peripheren Vollblut von 15 Seminompatienten durch, je 5 Patienten aus den drei Gruppen lymphogen metastasierter, okkult metastasierter und nicht metastasierter Seminome. Durchschnittlich fanden sich 1,3x107, 1,2x107 und 1,2x107 Reads bei den lymphogen, okkult und nicht metastasierten Seminomen. Nach dem „Trimming“ blieben durchschnittlich 73 - 76 % übrig, wovon 80 - 84 % annotierte Reads mit 7,2 - 7,8 % (1,6 - 1,7x104) annotierten small RNAs. Insgesamt zeigten 137 small RNAs eine Genexpression >2 und mindestens 50 Reads. In der univariaten Analyse konnten 35, 32 und 38 small RNAs identifiziert werden, die eine signifikante Diskriminierung zwischen lymphogen/okkult metastasiert bzw. beider metastasierter Subtypen kombiniert von den nicht metastasierten Seminomen ermöglichten (Abb. 4). Eine Differenzierung zwischen okkult und sichtbar metastasierten Seminomen war nicht möglich. Schließlich nutzten wir Support Vector Machine zur kompletten Diskriminierung metastasierter und nicht metastasierter Stadien. Insgesamt waren 891, 668 und 87 Kombinationen möglich, die eine vollständige Diskriminierung der lymphogen, okkult metastasierten und der insgesamt metastasierten Seminome von den nicht metastasierten Seminomen ermöglicht.
Als Ergebnis dieser Untersuchung fanden wir small RNAs, die einen molekularen Marker zur Differenzierung metastasierter und nicht metastasierter Seminome im peripheren Vollblut darstellen können. Anhand der Expression zweier small RNAs ist damit erstmals eine vollständige Diskriminierung metastasierter und nicht metastasierter Seminome im Vollblut möglich.
Auch bei den Ergebnissen der small RNA Expression im Blut zeigte sich, dass metastasierte Tumoren sich auf small RNA-Ebene von den nicht metastasierten unterscheiden, eine Unterscheidung der metastasierten untereinander aber nicht möglich ist. Dies bestätigt die bisherigen Erkenntnisse und die These der biologischen Gemeinsamkeit okkult und sichtbar metastasierter Tumoren. Die Validierung der Ergebnisse an einem unabhängigen größeren Kollektiv muss erfolgen, um die diagnostische Aussagekraft der small RNAs zu bestätigen.
Schlussfolgerungen
In unseren Studien wurde schrittweise und systematisch nach molekularen Markern zur Diskriminierung metastasierter und nicht metastasierter Seminome gesucht. Im ersten Schritt wurde die Expression der messenger RNAs [14, 15] und im zweiten Schritt die der small RNAs [20, 25, 26] untersucht. Zunächst verglichen wir die Genexpression im Primärtumor metastasierter und nicht metastasierter Seminome als dem Ort, an dem die biologischen Prozesse ablaufen. In einem weiteren Schritt untersuchten wir die small RNA Expression im Vollblut, um so eine bessere klinische Anwendbarkeit zu erreichen [25]. Zu Beginn der Untersuchungen war unklar, welche mRNAs oder small RNAs eine Diskriminierungsfähigkeit besitzen, sodass wir zunächst ein umfassendes Screening aller bekannten Gene und small RNAs mittels Whole Genome Mikroarray bzw. „small RNA Next Generation Sequencing“ durchführten.
Die Vorhersagbarkeit des Metastasierungsstatus bei Seminomen stieg mit jeder Untersuchung. Vor dem Jahr 2002 waren keine Risikofaktoren bekannt und die aera under the curve (AUC) zur Vorhersage einer Metastasierung lag damit bei 50 % (Abb. 5). Durch die Untersuchung von klinischen Risikofaktoren besserte sich die Konkordanz auf 65 %. Mit der Bestimmung der mRNA Expression als molekularer Marker steigerte sich die Konkordanz anhand der Expression zweier Gene (DRD1 und FAM72F1) auf 88 %. Das Ziel einer vollständigen (100 %) Differenzierung zwischen metastasierten und nicht metastasierten Seminomen wurde schließlich durch Untersuchung der Expression von small RNAs erreicht. Damit lagen erste Hinweise auf eine funktionierende Diagnostik/Prädiktion des Metastasierungsstatus sowohl an Material aus dem Primärtumor als auch an peripherem Vollblut vor.
Als zusätzlichen Schritt konnten wir nachweisen, dass sich okkult und sichtbar metastasierte Seminome auf transkriptionaler Ebene nicht unterscheiden. Die Validierung unserer Ergebnisse an einem größeren und unabhängigen Kollektiv befindet sich in der Planung. Sollte sie gelingen, könnte eine okkulte Metastasierung frühzeitig diagnostiziert und eine adjuvante Therapie oder eine intensivere Nachsorge eingeleitet werden. Dies würde die häufige klinische Entscheidung für oder gegen eine Therapie erleichtern. Nicht metastasierte Patienten bedürften keiner weiteren Therapie. Eine unnötige Exposition gegenüber potentiell toxischen Substanzen (Chemotherapie bzw. ionisierende Strahlung) und die damit verbundenen Langzeitschäden könnten so vermieden werden.
1 Mathematisches Verfahren zur Einteilung einer Menge von Objekten so in Klassen, dass um die jeweilige Klasse ein möglichst großer freier Raum, also Abstand, zu anderen Klassen besteht.
Literatur
- Ruf CG, Isbarn H, Wagner W, Fisch M, Matthies C, Dieckmann KP: Changes in epidemiologic features of testicular germ cell cancer: Age at diagnosis and relative frequency of seminoma are constantly and significantly increasing(). Urologic oncology 2013.
- Mette Sakso Mortensen MGG, Jakob Lauritsen, Mads Agerbaek, Niels Vilstrup Holm, Hans von der Maase, Gedske Daugaard: A nationwide cohort study of surveillance for stage I seminoma. J Clin Oncol 2013, J Clin Oncol 31, 2013 (suppl; abstr 4502).
- Warde P, Specht L, Horwich A, Oliver T, Panzarella T, Gospodarowicz M, von der Maase H: Prognostic factors for relapse in stage I seminoma managed by surveillance: a pooled analysis. J Clin Oncol 2002, 20(22):4448-4452.
- Chung P, Mayhew LA, Warde P, Winquist E, Lukka H, Genitourinary Cancer Disease Site Group of Cancer Care Ontario’s Program in Evidence-based C: Management of stage I seminomatous testicular cancer: a systematic review. Clinical oncology 2010, 22(1):6-16.
- Ruf CG, Khalili-Harbi N, Sachs S, Isbarn H, Wagner W, Matthies C, Meineke V, Fisch M, Chun FK, Abend M: The Search for Biomarkers of Metastatic Seminoma. The Journal of urology 2013.
- Valdevenito JP, Gallegos I, Fernandez C, Acevedo C, Palma R: Correlation between primary tumor pathologic features and presence of clinical metastasis at diagnosis of testicular seminoma. Urology 2007, 70(4):777-780.
- Kollmannsberger C, Tyldesley S, Moore C, Chi KN, Murray N, Daneshmand S, Black P, Duncan G, Hayes-Lattin B, Nichols C: Evolution in management of testicular seminoma: population-based outcomes with selective utilization of active therapies. Ann Oncol 2011, 22(4):808-814.
- Meinardi MT, Gietema JA, van der Graaf WT, van Veldhuisen DJ, Runne MA, Sluiter WJ, de Vries EG, Willemse PB, Mulder NH, van den Berg MP et al: Cardiovascular morbidity in long-term survivors of metastatic testicular cancer. J Clin Oncol 2000, 18(8):1725-1732.
- Oliver RT, Ong J, Shamash J, Ravi R, Nagund V, Harper P, Ostrowski MJ, Sizer B, Levay J, Robinson A et al: Long-term follow-up of Anglian Germ Cell Cancer Group surveillance versus patients with Stage 1 nonseminoma treated with adjuvant chemotherapy. Urology 2004, 63(3):556-561.
- Travis LB, Andersson M, Gospodarowicz M, van Leeuwen FE, Bergfeldt K, Lynch CF, Curtis RE, Kohler BA, Wiklund T, Storm H et al: Treatment-associated leukemia following testicular cancer. J Natl Cancer Inst 2000, 92(14):1165-1171.
Interessenkonflikt
Der Autor erklärt, dass kein Interessenkonfilkt im Sinne der Richtlinien des International Committee of Medical Journal Editors besteht.
Die Studie wurde durch Sonderforschungsgelder des Bundesministeriums der Verteidigung finanziell unterstützt.
Bildquelle: Abbildungen 1-5: Oberfeldarzt Dr. Ruf, Koblenz
Die Arbeit konnte nur durch die erfolgreiche Vernetzung der Bundeswehrkrankenhäuser und des Instituts für Radiobiologie der Bundeswehr entstehen. Mein Dank gilt daher allen, die auf Seiten der Krankenhäuser und des Instituts an dem Projekt mitgewirkt haben. Mein besonderer Dank gilt Herrn Oberstarzt Prof. Michael Abend und seiner Arbeitsgruppe „Genomics“ vom Insitut für Radiobiologie der Bundeswehr. Er hat mit seiner hervorragenden grundlagenwissenschaftlichen und epidemiologischen Expertise vom Studiendesign bis zur Veröffentlichung maßgeblich mitgewirkt.
Abb. 1: Whole Genome Mikroarray Chips der Firma Applied Biosystems (Life Technologies, Darmstadt)
Abb. 2: Amplifikationsplot als grafische Darstellung der Amplifikationsergebnisse für jedes Gen eines Laufes.
Abb. 3: Schnittmengendiagramm der Anzahl der small RNAs, die eine Differenzierung zwischen den okkult/lymphogen oder aller metastasierten Seminome kombiniert versus nicht metastasierten Seminomen ermöglicht.
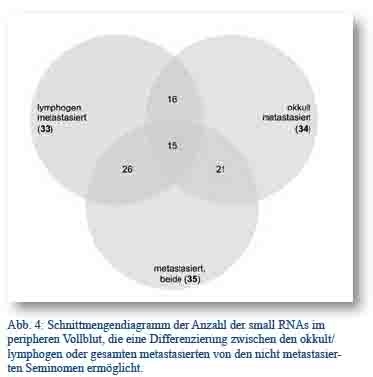
Abb. 4: Schnittmengendiagramm der Anzahl der small RNAs im peripheren Vollblut, die eine Differenzierung zwischen den okkult/ lymphogen oder gesamten metastasierten von den nicht metastasierten Seminomen ermöglicht.
Abb. 5: Grafische Darstellung der Vorhersagewahrscheinlichkeit (AUC) des Metastasierungsstatus bei Seminomen unter Nutzung klinischer und molekularbiologischer Marker.
Datum: 23.02.2015
Quelle: Wehrmedizinische Monatsschrift 2014/10-11