MIKROBIOLOGISCHE PCR-NACHWEISVERFAHREN IM EINSATZ*
Microbiological PCR Detection Methods in Missions*
Aus der Abteilung II - Veterinärmedizin (Abteilungsleiter: Oberfeldveterinär Dr. N. Langfeldt) am Zentralen Institut des Sanitätsdienstes der Bundeswehr Kiel (Institutsleiter: Oberstveterinär Dr. H.-H. Pott)
Henrik Tandler, Ulrich Schotte und Alfred Binder
Molekularbiologische Nachweisverfahren haben in allen Bereichen der mikrobiologischen Diagnostik zu enormen Fortschritten geführt. Insbesondere für virologische Nachweise ist die PCR das einzige praktikable Detektionsverfahren im Routinelabor. Im März 2011 wurde die Real time-PCR als hochsensibles Nachweisverfahren für Tollwut-, Schweinepest- sowie Noroviren und aus gegebenem Anlass für EHEC im veterinärmedizinischen Labor (VetMedLab) des Sanitätseinsatzverbandes (SanEinsVbd) bei KFOR eingeführt und im Auslandseinsatz etabliert.
Proben wurden u. a. in Zusammenarbeit mit der Food and Veterinary Agency in Pristina, in die das Food and Veterinary Laboratory als Referenzlabor für veterinärmedizinische Untersuchungen im Kosovo eingegliedert ist, untersucht. Die Geräteausstattung, bestehend aus Schüttelmühle zum Aufschluss der Organproben, EZ1-Aufreinigungsautomat für die standardisierte Aufbereitung von RNA sowie DNA und dem SMART-Cycler als robusten und kompakten PCR-Thermocycler für den Labor- bzw. Containereinsatz, wurde speziell für den Einsatz zusammengestellt und die Nachweisverfahren auf diese Geräte angepasst. Mit über 1 100 PCR-Nachweisen konnte die universelle Nutzung der PCR-Ausstattung, die sowohl für die Veterinärmedizinischen Einsatzlabore im Containermodul wie auch in fester Infrastruktur geeignet ist, in allen Kriterien der molekularbiologischen Diagnostik bestätigt werden. Die Erprobung der Geräteausstattung und der Verfahren erfolgte im Regeleinsatz bei KFOR über inzwischen 16 Monate mit Integration der Methoden in den Routinelaborbetrieb und häufigem Personalwechsel. Die Koordination der Abläufe unter Berücksichtigung der Probenbearbeitung aus den Bereichen Trinkwasser, Lebensmittel, Hygienestatuskontrollen sowie der Untersuchung von Proben von Diensthunden verlief problemlos.
Summary
Molecular detection methods have resulted in tremendous advances in all fields of diagnostic microbiology. Especially for virus detection PCR became the most feasible method in routine laboratories. Since March 2011 Real time PCR was implemented in the Laboratory for Veterinary Medicine of the KFOR Medical Task Force for the highly sensitive detection of rabies, classical swine-fever, noroviruses and EHEC relating to last year's outbreak in Germany. Some samples for the detection of animal infectious diseases and zoonoses were investigated in cooperation with the Food and Veterinary Laboratory as the reference laboratory for veterinary diagnostics in Kosovo, which is part of the Food and Veterinary Agency in Pristina. The laboratory equipment consisted of a bead mill, the EZ 1-workstation for standardised DNA-RNA extraction and the SMART-Cycler. The robust and compact design was especially selected for out of area missions to be used in local facilities or laboratory containers. Validated standard protocols were adapted to the equipment. Until now, more than 1100 PCR-runs met all criteria for veterinary molecular diagnostic systems and thus confirmed the universal usability of this system. The trial succeeded over 16 months in a working routine laboratory in spite of regular staff changes. No problems occurred in the close coordination with the sample processes for water, food, hygiene-control or samples from military dogs.
1. Einführung
In den Abteilungen Veterinärmedizin der Zentralinstitute des Sanitätsdienstes der Bundeswehr werden im Bereich Lebensmittelhygiene und gesundheitlicher Verbraucherschutz regelmäßig Lebensmittel, Trinkwasser und Umgebungsproben untersucht. Im Schwerpunktinstitut Veterinärmedizin in Kiel-Kronshagen wird zusätzlich Tierseuchen- und Zoonosendiagnostik durchgeführt.
Mit der Einführung von umfangreichen kulturellen und in jüngerer Vergangenheit auch molekularbiologischen Methoden kann in den veterinärmedizinischen Untersuchungseinrichtungen der Bundeswehr die mikrobiologische Diagnostik mit hohem Qualitätsstandard durchgeführt werden. Durch die Etablierung von PCR-Nachweisen im VetMedLab konnte für den Auslandseinsatz das Spektrum der diagnostischen Möglichkeiten erheblich erweitert werden.
Im Rahmen des Sonderforschungsvorhabens „Nachweis des Tollwutvirus nach automatisierter RNA-Präparation aus Organen mittels Real time-RT-PCR“ am ZInstSanBw Kiel wurde die Erprobung der Real time-RT-PCR als schnelles hochsensitives Verfahren im Veterinärmedizinischen Einsatzlabor im 28. Kontingent KFOR ab März 2011 eingeführt und bis heute fortgesetzt.
Dabei erfolgte die Überprüfung der Praktikabilität, der Funktion und der universellen Nutzung der Geräteausstattung und der Testmethoden. Neben der Tollwut-PCR wurden auch Nachweisverfahren für Noroviren, Schweinepest und aus aktuellem Anlass für enterohämorrhagische Escherichia coli (EHEC) etabliert und im Einsatz durchgeführt.
2. Methoden und Ergebnisse
Der molekularbiologische Nachweis von RNA-Viren (Tollwut-Virus, Virus der Klassichen Schweinepest, Norovirus) erfolgte mittels Real time-RT-PCR nach Elution der Viren aus dem Probenmaterial und automatisierter Präparation der RNA über den EZ1-Aufreinigungsautomaten. Die RNA muss für die PCR mit Reverser Transkriptase in DNA umgeschrieben werden, daher die Bezeichnung RT-PCR. Diese Zusammenstellung ermöglicht die PCR-Diagnostik im Einsatz sowohl im Verbund mit dem Veterinärmedizinischen Laborcontainer (VML) als auch in fester Infrastruktur.
Die Geräteausstattung (Abb 1), bestehend aus
- Schüttelmühle (Fa. Qiagen) zur Zerkleinerung und zum Aufschluss der Organproben,
- EZ1-Aufreinigungsautomat (Fa. Qiagen) für die standardisierte und rationelle Aufbereitung von RNA sowie DNA und
- SMART-Cycler (Fa. Cepheid) als robuster und kompakter PCR-Thermocycler für den Labor- bzw. Containereinsatz,
wurde speziell für den Einsatz zusammengestellt und die einzelnen Nachweisverfahren auf diese angepasst.
Der Aufbau, die Inbetriebnahme und die Nutzung der Geräte im Veterinärmedizinischen Einsatzlabor der Klinik SanEinsVbd in Prizren gestaltete sich problemlos.
Während des Berichtszeitraumes wurde für den stationären Einsatz in fester Infrastruktur ein zusätzlicher Raum für die kontaminationsfreie Präparation der Reagenzien einschließlich Master-Mix und Primer-Sonden-Ansätzen im Klinikbereich zur Verfügung gestellt (Abb 2). Weitere spezielle Laborgeräte waren hier nicht erforderlich.
Das Verbrauchsmaterial und auch spezielle Reagenzien für die Durchführung der Real time-RT-PCR und die Aufreinigung von Nukleinsäuren wurden vollständig über etablierte logistische Verfahren an das VetMedLab im Einsatz (i. E.) geliefert. Auch die Lieferung von tiefgefrorenen Reagenzien wurde ohne Probleme abgewickelt.
Die Geräte und insbesondere empfindliche Reagenzien waren im Einsatzlabor über den bisherigen Zeitraum von 16 Monaten voll funktionsfähig. Eingesetztes Laborpersonal wurde im Vorfeld in die für den Einsatz etablierte Diagnostik eingewiesen, sodass es trotz Personalwechsel nicht zu Problemen bei der Durchführung kam. Durch den Einsatz automatisierter RNA-Präparation und gebrauchsfertig aliquotierter Reagenzien wurden personalbedingte Einflüsse auf die Qualität der Ergebnisse zudem deutlich reduziert.
Untersuchungen zur Tollwut und Schweinepest erfolgten in Zusammenarbeit mit der Food and Veterinary Agency (FVA, Abb 3) in Pristina, die als Untereinheit das Food and Veterinary Laboratory als derzeit einzig gut ausgestattetes Veterinärlabor im Kosovo betreibt. Die FVA erhält unter anderem im Rahmen eines sogenannten „Twinning“-Projektes der EU, bei dem das deutsche Bundesministerium für Ernährung, Landwirschaft und Verbraucherschutz federführend ist, Unterstützung in allen relevanten Fragestellungen durch deutsche Fachexperten, beispielsweise vom Niedersächsischen Landesamt für Verbraucherschutz und Lebensmittelsicherheit (LAVES) sowie vom Friedrich-Loeffler-Institut (Insel Riems). Ziel des Twinnings ist es, die Kapazitäten der Agentur zu stärken und ein entsprechendes Kontrollsystem aufzubauen, um somit die Übernahme und Anwendung des EU-Rechts im Bereich der tierischen und pflanzlichen Gesundheit zu ermöglichen.
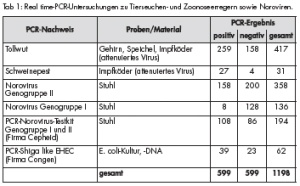
In Tabelle 1 ist eine Übersicht aller PCR-Ergebnisse zusammengestellt. Zur Methodenetablierung wurde Proben- und Referenzmaterial teilweise mehrfach untersucht. Daher ist die Zahl der Untersuchungen deutlich höher als die Zahl der Proben.
2.1. Tollwut
Die Tollwut ist eine weltweit verbreitete Zoonose, der nach Schätzung der WHO jährlich etwa 55 000 Menschen mit Schwerpunkt in Asien und Afrika zum Opfer fallen; die Dunkelziffer ist durch mangelhaftes Meldewesen hoch einzuschätzen [1]. In 2011 erkrankte ein US-Soldat nach einem Hundebiss in Afghanistan an der Tollwut. Nach der Verletzung im Januar hielt er sich in Deutschland und den USA auf, wurde mehrfach ärztlich behandelt, ohne dass an eine Tollwutprophylaxe gedacht wurde, und starb schließlich 7 Monate nach Infektion im August 2011 [2]. Die hohe Letalität, verbunden mit dem endemischen Vorkommen in derzeitigen und möglicherweise zukünftigen Einsatzländern, erfordert eine feldtaugliche zuverlässige Diagnostik dieser Viruserkrankung. Der Goldstandard zum Nachweis des Tollwut-Virus post mortem ist die direkte Immunfluoreszenz in fixierten Tupfpräparaten verschiedener Hirnabschnitte [3]. Da die Sicherheit dieser Diagnostik sehr von der Erfahrung des Untersuchers sowie dem Zustand und der Qualität des Untersuchungsmaterials abhängig ist, wurde für den Auslandseinsatz im Rahmen eines Sonderforschungsvorhabens* eine Real time-RT-PCR als zweites, hoch sensitives und spezifisches Tollwut-Nachweissystem etabliert [4, 5]. Dieses Verfahren hat den Vorteil, dass auch stark autolytisches Hirnmaterial untersucht werden kann (Abb 4).
Im Untersuchungszeitraum wurden zur Diagnostik der Tollwut vier Gehirnproben (zwei positive Proben von Hunden aus der FVA, 2 negative Proben von Rindern), eine Speichelprobe eines streunenden Hundes aus dem Feldlager Prizren und 6 Tollwut-Impfköder untersucht. Letztere stammen aus der von der Europäischen Union auf dem ganzen Balkan durchgeführten aktiven Immunisierung gegen die Tollwut in der Wildtierpopulation. Im Camp Bondsteel wurden die US-amerikanischen Kollegen durch den Leitenden Veterinär, den Leiter VetMedLab und den Gesundheitsaufseher im Einsatz bei der Ausbringung der Impfköder unterstützt (Abb 5).
2.2. Schweinepest
Bei der klassischen Schweinepest handelt es sich um eine hoch ansteckende, fieberhaft verlaufende Viruserkrankung die sowohl Haus – als auch Wildschweine befällt. Die Schweinepest ist aufgrund ihrer hohen wirtschaftlichen Bedeutung national und international anzeigepflichtig. Bei einem Ausbruch dieser Tierseuche muss der gesamte Tierbestand aus Gründen der hohen Infektiosität getötet werden. Eine Impfung von Hausschweinen ist in der EU derzeit verboten. Daher liegt der Schwerpunkt auf der Vorbeugung und Überwachung dieser Tierseuche. Wildschweine und von ihnen ausgehende Kontaminationswege stellen in Europa zunehmend die wichtigste Infektionsquelle für Hausschweinbestände dar. Daher ist das Monitoring der Schwarzwildbestände auf Schweinepest-Viren und eng verwandte Viren von zentraler Bedeutung [6, 7].
In Zusammenarbeit mit der FVA, die auch für die Tierseuchenbekämpfung im Kosovo zuständig ist, wurden aus zwei Schweinepestimpfstoffen verschiedener Hersteller 26 RNA-Aufreinigungen gewonnen und insgesamt 31 Untersuchungen mittels Real time-RT-PCR durchgeführt. Dabei wurde festgestellt, dass sich die beiden Impfstoffe hinsichtlich des Virustiters stark unterscheiden. Daher ist bei einem Impfstoff mit einer deutlich reduzierten immunogenen Wirkung und einem damit verbundenen unsicheren Impfstatus der geimpften Tiere zu rechnen.
2.3. Norovirus
Noroviren gelten weltweit als die wichtigsten Verursacher von akuten viralen Gastroenteritiden. Nach heutigen Erkenntnissen ist der Mensch als einziger Träger dieses Virus bekannt. Minimale Mengen von 10-100 Viruspartikeln reichen für eine Infektion aus. Das Virus ist ganzjährig im Umlauf, wobei der Schwerpunkt der Infektionen in den Wintermonaten liegt. Da es nicht möglich ist, das Virus in Zellkultur anzuzüchten, gilt die Real time-RT-PCR als Goldstandard zur Detektion von Noroviren aus Stuhl-, Lebensmittel- oder Oberflächenproben. Bei Soldaten im Auslandseinsatz zählen Durchfallerkrankungen zu den häufigsten Ausfallursachen. So berichten beispielsweise Amerikaner und Franzosen aus ihren Einsatzgebieten regelmäßig von durch Noroviren bedingten Durchfallepidemien [8, 9]. Durch den begrenzten Raum in den Feldlagern und die große Stabilität des Virus gegenüber Desinfektionsmitteln und Umwelteinflüssen ist ein rechtzeitiger und genauer Nachweis unerlässlich, um bereits in der ersten Phase der Ausbruchsbekämpfung eine weitere Ausbreitung der Infektion unter den Soldaten zu verhindern.
Für den Einsatz im In- und Ausland geeignete Nachweissysteme für Noroviren aus unterschiedlichen Probenmatrices wurden im Rahmen eines Forschungsvorhabens* durch das Institut für Lebensmittelqualität und -sicherheit der Tierärztlichen Hochschule Hannover etabliert und standardisiert.
Die PCR-Nachweise erfolgten mit verschiedenen Protokollen. Zur Detektion der Genogruppe I (GG I) wurde das Protokoll nach da Silva et al. [11] und für die Genogruppe II (GG II) das Protokoll nach Höhne und Schreier [12] angewendet. Des Weiteren wurde das RT-PCR-Protokoll des Cepheid-Testkits genutzt, welches in einem Schritt sowohl GG I als auch GG II nachweist.
Ende Juni 2011 kam es in einem Camp anderer KFOR-Partner immer wieder zu Durchfallerkrankungen mit etwa 2 bis 4 betroffenen Soldaten pro Tag. Nachdem zunächst weder eine bakteriologische noch eine virologische Ursache für die Erkrankung nachgewiesen werden konnte, wurden in diesem Zusammenhang Stuhlproben von zwei Patienten mittels Real time-RT-PCR im VetMedLab i. E. untersucht. Dadurch ließ sich zweifelsfrei eine Infektion mit Noroviren nachweisen. Bis Ende 2011 wurden 82 Stuhlproben von Durchfallpatienten auf Noroviren untersucht. In 39 Fällen wurde eindeutig eine Infektion mit Noroviren nachgewiesen. Zur Etablierung der Real time-RT-PCR im Einsatz wurden die Stuhlproben wiederholt aufgereinigt und mittels verschiedener Protokolle untersucht. So konnten 34 Infektionen mit Norovirus Genogruppe II, drei Infektionen mit der in unseren Breiten seltenen Genogruppe I und zwei seltene Doppelinfektionen mit sowohl Genogruppe I als auch II nachgewiesen werden. Mit den Daten von Donia et al. [10] handelt es sich hier um einen der ersten Berichte zum Vorkommen von Noroviren auf dem Balkan und die erste Beschreibung im Kosovo.
Vergleichende Untersuchungen zwischen dem VetMedLab i. E. und ZInstSanBw Kiel der Eluate und der Proben ergaben eine gute Vergleichbarkeit der Ergebnisse. Es traten weder falsch positive noch falsch negative Proben auf, auch die ct-Werte im Vergleich ergaben in der Summe nur geringe Abweichungen.
2.4. EHEC
EHEC (enterohämorrhagische E. coli) zeichnen sich durch diverse Virulenzfaktoren aus, wobei alle EHEC-Stämme über das Phagen-kodierte Shiga-Toxin als Hauptvirulenzfaktor verfügen. Als Zoonoseerreger werden EHEC vornehmlich über Lebensmittel aber in Einzelfällen auch durch Schmierinfektionen auf den Menschen übertragen. Das klinische Erscheinungsbild variiert zwischen milden gastrointestinalen Symptomen bis hin zu lebensbedrohlichen und zum Teil letalen systemischen Erkrankungen mit Nierenbeteiligung, in Form des hämolytisch-urämischen Syndroms (HUS). In den Monaten Mai bis Juli 2011 kam es in Deutschland zu zahlreichen schwerwiegenden Infektionen mit EHEC des bis dahin unbekannten Serotyps O104:H4 (EHEC O104) [13,14]. Im Zuge dieser lebensmittelbedingten Epidemie wurden dem VetMedLabor i. E. KFOR durch das ZInstSanBw Kiel kurzfristig als Präventivmaßnahme ein Real time-PCR-Protokoll und die dazugehörigen Reagenzien (PCR-Testkit der Fa. Congen) zur Verfügung gestellt. Als Kontrolle diente Referenzmaterial eines inaktivierten EHEC-Stammes. Zur Etablierung der Methode im Einsatz wurden DNA-Eluate direkt untersucht oder E. coli-Stämme von einer Blutagarplatte oder aus einer Anreicherungsbouillon mit DNA-Eluat gespikt. Im Zuge der Untersuchungen wurden 22 DNA-Aufreinigungen und 62 PCR-Untersuchungen durchgeführt. In allen Untersuchungen wurden die EHEC-Erreger identifiziert.
3. Diskussion
Nach Entwicklung der PCR vor etwa 25 Jahren hat seit 10 Jahren die Real time-PCR insbesondere für diagnostische Verfahren ungeahnte Möglichkeiten eröffnet. In der konventionellen PCR erfolgt die Auswertung der Amplifikate nach Abschluss der PCR in einer Elektrophorese. Dieser Ablauf ist nicht nur zeit- und arbeitsaufwändig sondern auch durch offene Manipulation der massenhaft vervielfältigten Amplifikate die größte Gefahrenquelle für Kontaminationen. In der Real time-PCR wird neben den Primern für die Vervielfältigung der DNA eine Sonde mit fluoreszierendem Farbstoff verwendet. Die Sonde bindet in der PCR nur dann, wenn sie zum amplifizierten DNA-Abschnitt passt, und ist damit ein wichtiges zusätzliches Kriterium für die Spezifität des Tests. Außerdem kann durch den an die Sonde gebundenen fluoreszierenden Farbstoff das Ergebnis der PCR während der Reaktion ausgewertet werden (real time). Der Ablauf wird damit erheblich beschleunigt. Durch die eingesetzte Markierung der Sonde ist das Real time-Format deutlich sensitiver als eine herkömmliche PCR mit anschließender Gelelektrophorese. Nach Abschluss des Tests können die geschlossenen Reaktionsgefäße entsorgt werden. Dadurch wird eine wesentliche Kontaminationsquelle im Laborablauf vermieden. Zudem eignet sich diese Technik auch für die Untersuchung großer Probenzahlen.
Der Aufreinigungsautomat EZ1 führt die Aufreinigung standardisiert unter entsprechenden Qualitätskriterien durch. Er ist universell für die Aufarbeitung von Material zum Nachweis von Viren, Bakterien und Parasiten geeignet. Die manuelle Aufreinigung erfordert dagegen zahlreiche zeit- und arbeitsintensive Zentrifugationsschritte, die das Personal binden, und ist daher im Einsatzlabor nur als Alternative bei Ausfall des Automaten geeignet. Die automatisierte Aufreinigung reduziert zudem erheblich mögliche Kontaminationen, deren Risiko bei manuellen Arbeiten deutlich erhöht ist.
Zusammenfassend kann festgestellt werden, dass das etablierte System sich im Einsatz sowohl unter schwierigeren räumlichen Bedingungen, trotz häufiger Personalwechsel und trotz hoher Anforderungen an die Flexibilität bewährt hat. Die Methoden zur Vorbereitung der Proben und die automatisierte Aufreinigung von wahlweise DNA und/oder RNA ermöglicht ein außergewöhnlich breites Einsatzspektrum und kann jederzeit an aktuelle Fragestellungen angepasst werden. Da im Bereich der Veterinärmedizin Etablierung und Validierung der diagnostischen Systeme am Schwerpunktinstitut durchgeführt beziehungsweise koordiniert werden, kann grundsätzlich für jedes Einsatzszenario die Diagnostik im Einsatz einschließlich zusätzlicher bestätigender Diagnostik im Inland vorgehalten werden.
4. Schlussfolgerungen
Im Hinblick auf eine universelle Nutzung der Ausstattung für Veterinärmedizinische Einsatzlabore im Containermodul wie auch in fester Infrastruktur hat sich die PCR-Ausstattung in allen Kriterien für die molekularbiologische Diagnostik bewährt:
- Lieferung, Lagerung und Nutzung empfindlicher Reagenzien, die zum Teil tiefgefroren gelagert werden müssen, ist problemlos möglich.
- Für die Isolierung und Bearbeitung der empfindlichen RNA oder DNA ist der EZ1-DNA-/RNA-Aufschlussautomat auch bei beengten räumlichen Verhältnissen optimal geeignet.
- Die Real time-PCR im SMART-Cycler bietet den großen Vorteil der Ergebnisauswertung bereits im PCR-Lauf. Nach Abschluss des Tests erfolgt die Auswertung des Sondensignals für positive oder negative Reaktionen. Eine arbeitsintensive Aufarbeitung der Amplifikate für die Gelelektrophorese entfällt. Außerdem müssen die Amplifikate nicht weiter verarbeitet werden, sodass das Kontaminationsrisiko minimiert wird.
- Am Beispiel des EHEC-PCR-Nachweises konnte gezeigt werden, dass mit der im Einsatz bei KFOR zur Verfügung gestellten Ausstattung sehr schnell aktuelle diagnostische Fragestellungen bearbeitet werden können. Voraussetzung hierfür ist, dass im Inland bereits etablierte Protokolle vorliegen, die verzugslos mit den Geräten im Einsatz durchgeführt werden können.
- Trotz des einsatzbedingten häufigen Personalwechsels konnte nach vorheriger Einweisung des Personals die Diagnostik ohne Einschränkungen weitergeführt werden.
Die Erprobung der Geräteausstattung und der Verfahren erfolgte im Regeleinsatz bei KFOR mit Integration der Methoden in den Routine-Laborbetrieb. Die Koordination der Abläufe unter Berücksichtigung der Probenbearbeitung aus den Bereichen Trinkwasser, Lebensmittel und Hygienestatuskontrollen sowie der Untersuchung von Proben von Diensthunden verlief reibungslos. Obgleich das VetMedLab inzwischen den veterinärmedizinischen Anteil des Theatre Preventive Medical Laboratory für die NATO stellt und somit zusätzliche Untersuchungen mit umfangreichem Probeneingang aus zahlreichen Feldlagern im Kosovo zu bearbeiten hat, konnten mit der dargestellten Methode zeitnah sichere Ergebnisse erzielt werden.
Literatur
- World Health Organization (WHO). Human and animal rabies. http://www.who.int/rabies/en/
- Cersovsky A, Rupprecht CE, Maxted AM, Lankau EW: Imported Human Rabies in a U.S. Army Soldier – New York, 2011. Morbidity and Mortality Weekly Report 2012; 61 (17): 302-305.
- World Organization for Animal Health [OIE]. Manual of diagnostic tests and vaccines for terrestrial animals. OIE; 2008. Rabies. http://www.oie.int/fileadmin/Home/eng/Health_standards/tahm/2.01.13_RABIES.pdf
- Hoffmann B, Freuling CM, Wakeley PR, Rasmussen TB, Leech S, Fooks AR, et al.: Improved safety for molecular diagnosis of classical rabies viruses by use of a TaqMan real-time reverse transcription-PCR "double check" strategy. J Clin Microbiol 2010: 3970-3978.
- Binder A, Teifke JP, Saturday GA, Hoffmann B, Freuling C, Müller T, Sauer S: Tollwut – Entwicklung einsatzorientierter Diagnostikverfahren. Wehrmed Monatsschr 2010; 290-292.
- Hoffmann B, Beer M, Schelp C, Schirrmeier H, Depner K: Validation of a Real-Time RT-PCR assay for sensitive and specific detection of classical swine fever. J Virol Methods 2005; 36-44.
- Leifer I, Hoffmann B, Höper D, Bruun Rasmussen T, Blome S, Strebelow G, et al.: Molecular epidemiology of current classical swine fever virus isolates of wild boar in Germany. J Gen Virol 2010; 91: 2687-2697.
- Mayet A, Andreo V, Bedubourg G, Victorion S, Plantec J, Soullie B et al.: Food-borne outbreak of norovirus infection in a French military parachuting unit, April 2011. Euro Surveill 2011; 16: 30.
- Thornton SA, Sherman SS, Farkas T, Zhong W, Torres P, Jiang X: Gastroenteritis in US Marines during Operation Iraqi Freedom. Clin Infect Dis 2005; 40 (4): 519-525.
- Donia, D, Kota, M, Leno, L, Ylli, A, Cenk, F, Divizia, M: First outbreak of norovirus in Albania. Lett Appl Microbiol 2011; 7: 283-287.
- Da Silva AK, Le Saux JC, Parnaudeau S, Pommepuy M, Elimelech M, Le Guyader FS: Evaluation of removal of noroviruses during wastewater treatment, using real-time reverse transcription-PCR: different behaviors of genogroups I and II. Appl Environm Microbiol 2007; 73: 7891-7897.
- Höhne M, Schreier E: Detection and Characterization of Norovirus Outbreaks in Germany: Application of a One-Tube RT-PCR Using a Fluorogenic Real-Time Detection System. J Med Virol 2004; 72: 312-319.
- Martin A: Outbreak of Shiga toxin-producing Escherichia coli (STEC) O104:H4 infection in Germany causes a paradigm shift with regard to human pathogenicity of STEC strains. J Food Protection 2012; 408-418.
- Muniesa M, Hammerl J A, Hertwig S, Appel B, Brüssow H: Shiga Toxin-Producing Escherichia coli O104:H4: A New Challenge for Microbiology. Appl Environm Microbiol 2012; 78: 4065-4073.
Fotos: Zentrales Institut des Sanitätsdienstes der Bundeswehr, KIEL.
* Entstanden im Rahmen von Forschungsvorhaben für den Sanitätsdienst der Bundeswehr (Projektnummer M/SAB1/7/A001, Tierärztliche Hochschule Hannover, Institut für Lebensmittelqualität und -sicherheit und Projektnummer 01Z5-S-850708, ZInstSanBw Kiel, Abteilung II Veterinärmedizin).
Datum: 20.09.2012
Wehrmedizinische Monatsschrift 2012/8-9