Etablierung einer genexpressionsbasierten Früh- und Hochdurchsatzdiagnostik der hämatologischen akuten Strahlenkrankheit
Matthäus Majewski
Aus der Arbeitsgruppe „Genomics“ (Arbeitsgruppenleiter: Oberstarzt Prof. Dr. M. Abend) des Instituts für Radiobiologe der Bundeswehr (Institutsleiter: Oberstarzt Prof Dr. M. Port)
Zusammenfassung
Einführung: Radiologische und nukleare (R/N) Szenarien sind potentielle Großschadenslagen, die bei mehreren hunderten und tausenden betroffenen Personen zur Ausbildung einer hämatologischen akuten Strahlenkrankheit (HASK) führen können. Nach einem solchen Ereignis müssen so früh wie möglich vermeintlich bestrahlte („worried wells“) sowie niedrig und stark strahlenexponierte Menschen identifiziert werden.
Material und Methoden: Bei 18 Pavianen (Papio anubis) eines bei unserem französischen Partnerinstitut (Institut de Recherche Biomédicale des Armées (IRBA)) bereits laufenden Tierexperiments[1] wurden im Sinne eines „predictive assays“ Vollblutproben vor und ein sowie zwei Tage nach Teil- bzw. Ganzkörperbestrahlung – Wochen, bevor die Tiere an der akuten Strahlenkrankheit erkrankten – entnommen und dem Institut für Radiobiologie der Bundeswehr -(InstRadBioBw) zur Verfügung gestellt. Auf Grundlage der Blutbildveränderungen (7 bis 203 Tage nach Bestrahlung) wurde der Schweregrad der sich später entwickelnden HASK diagnostiziert.
Über verschiedene Schritte wurden genexpressionsbasierte Marker mittels Ganz-Genome Mikroarrayanalyse und quantitativer real-time PCR im Blut identifiziert und in klinischen Modellen validiert.
Ergebnisse: Von insgesamt 866 potentiellen Genen wurden die 89 besten Kandidaten herausgearbeitet, die eine Identifizierung der HASK – innerhalb der ersten beiden Tage nach Bestrahlung – ermöglichten. 29 Gene ließen sich in einer unabhängigen und methodischen Validierung bestätigen. Insgesamt neun Gene wurden erfolgreich an den Vollblutproben von strahlenexponierten Patienten validiert (Übertragung auf ein klinisches Modell). Vorläufige Ergebnisse des Versuchs zur Früh- und Hochdurchsatzdiagnostik bestätigen, dass durch Verwendung der identifizierten Kandidatengene der Durchsatz der aktuellen Diagnostik von zehn Proben innerhalb von 3 bis 4 Tagen (etablierte Verfahren) auf 1 000 Proben innerhalb von 24 Stunden (neues genexpressionsbasiertes Verfahren) erhöht werden kann.
Schlussfolgerungen: In jahrelangen, aufwändigen Untersuchungen, die ihren Beginn in einer NATO-Kooperation mit unserem französischen Schwesterinstitut in einem dort bereits laufenden Tierexperiment hatten, konnte eine strahleninduzierte Gensignatur identifiziert werden. Diese Gensignatur erlaubt innerhalb der ersten beiden Tage nach Bestrahlung eine Vorhersage der später auftretenden akuten Strahlenkrankheit. Damit können Patientinnen und Patienten, die später lebensgefährlich erkranken, frühzeitig hospitalisiert und einer optimalen Therapie zugeführt werden. Dies führt zu einer entscheidend verbesserten Prognose. In einer Reihe weiterer Experimente wurden die Ergebnisse validiert und ihr Potential als Hochdurchsatzdiagnostik gegenüber etablierten Verfahren herausgearbeitet. Diese Arbeiten bilden somit einen wichtigen Baustein zur Schließung der wehrmedizinischen Fähigkeitslücke der Früh- und Hochdurchsatzdiagnostik der HASK.
Schlüsselwörter: Genexpression, hämatologischer Strahlenschaden, Früh- und Hochdurchsatzdiagnostik, HASK
Summary
Introduction: Radiological and nuclear scenarios (R/N) are potential mass casualty situations with hundreds to thousands of people affected, developing a hematological acute radiation syndrome (HARS). An early medical management is needed to distinguish between people who only think of being affected (so called “worried wells”) and those actually exposed to a low or high radiation dose.
Material and Methods: In 2014 blood samples from an already ongoing experiment were offered to our institute by the French Institut de Recherche Biomédicale des Armées (IRBA) as part of a French-German NATO-collaboration. 18 baboons (Papio anubis) were partially and whole-body irradiated. As predictive assay whole-blood samples were taken before and 1-2 days after irradiation, weeks before the animals developed the acute radiation syndrome. Later on, the grading of the HARS was determined based on blood cell counts 7-203 days after irradiation. In these blood samples different gene expression changes were detected by whole genome microarray analysis and quantitative real-time PCR and vali-dated against clinical models.
Results: From overall 866 potential genes the 89 best candidates for the identification of HARS during the first two days after irradiation were selected. Of these 29 were validated independently and methodically. A total of 9 genes were successfully validated on patient samples.
Preliminary results confirm the applicability of this method as an early- and high throughput diagnostic tool which allows the processing of up to 1.000 samples within 24 h compared to 10 samples being analyzed within 3-4 days by using standard diagnostic procedures.
Conclusions: In a several year spanning series of experiments that started as a NATO-cooperation with our French partner institute based on material of an already running animal experiment we were able to identify a signature of radiation responsive genes. This signature allows an early prediction of the grade of a later developed HARS within the first two days after a radiation exposure. Hereby, patients exposed to a life-threatening dose of ionizing radiation might be hospitalized and treated faster – resulting in a realistic chance of a better medical outcome. Our results were validated in a clinical model and turned out to be useful as high-throughput diagnostic. Therefore, this work might help to close a skill gap in an important matter of military medicine: finding an early and high-throughput diagnostic tool for the prediction of HARS.
Keywords: gene expression, hematological acute radiation syndrome, HARS, high-throughput diagnostic
Einleitung
Hintergrund

R/N Szenarien umfassen neben Reaktorunglücksfällen auch den terroristischen oder militärischen Einsatz von Radionukliden oder Kernwaffen. 2009 wurde durch den damaligen US-Präsidenten Barack Obama der Nuklearterrorismus als eine der zentralen Bedrohungen für die internationale Sicherheit herausgestellt. Auf den „Nuklearen Sicherheitsgipfeln“ [2] wurde seitdem die zunehmende Gefährdung durch nuklearen Terrorismus diskutiert und es wurden Maßnahmen erarbeitet, um die Anwendung von Strahlenquellen als Waffe zu verhindern.
Die missbräuchliche Nutzung von Radionukliden durch Terroristen ist in vielfältiger Form vorstellbar. Angefangen bei der Nutzung als Gift wie im Fall Litvinenko [5] oder aber beim Einsatz als Radiological Exposure Devices (REDs) [6].
Szenarien, die zu Großschadensereignissen führen, sind leicht vorstellbar. Beim Einsatz einer sogenannten „schmutzigen Bombe“ werden Radionuklide mit einem konventionellen Sprengkörper vermengt. Dass selbst geringe Mengen an Radionukliden enorme Auswirkungen haben können, zeigte der Unfall von Goiânia (Brasilien) 1987. Nur 93 g Caesium-137 bewirkten, dass mehr als 100 000 Personen auf eine Kontamination untersucht werden mussten. Mehrere hundert Menschen wurden tatsächlich zum Teil stark radioaktiv kontaminiert und es kam zu vier Todesfällen [7].
Noch weitreichender könnten die Folgen eines terroristischen Angriffs auf ein Kernkraftwerk sein. Dabei sind sowohl „konventionelle“ Attacken (Angriffe mittels Verkehrsflugzeug oder Überfall) als auch Cyberangriffe – wie im Fall Iran und Stuxnet – vorstellbar [8].
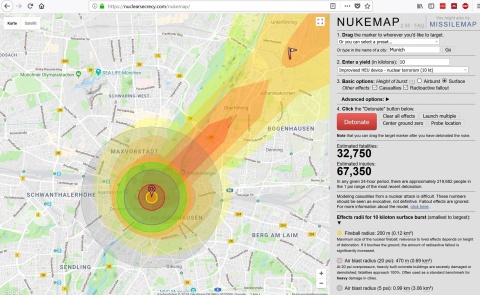
Das „worst-case Szenario“ einer nuklearen terroristischen Bedrohung ist der Einsatz einer Kernwaffe. Bei der Oberflächenzündung einer improvisierten Kernwaffe (Improvised Nuclear Device, IND) in einem Ballungszentrum ist mit verheerenden Folgen zu rechnen [9-11]. Selbst bei der Detonation einer vergleichsweise kleinen Bombe mit 10 Kilotonnen Sprengkraft ist mit mehreren zehntausend Betroffenen und der Verstrahlung großer Areale zu rechnen (Abbildung 1).
Dass die missbräuchliche Aneignung von Radionukliden weniger abwegig ist als man zunächst vermuten mag, zeigt die „Incident and Trafficking Database“ (ITDB) der internationalen Atomenergiebehörde (IAEA) (12). Diese Datenbank dokumentiert tausendfach den unerlaubten Handel und die Nutzung sowie den Verlust von Strahlenquellen.
Über die Aspekte Unglücksfälle und Terrorismus hinaus ist auch ein möglicher militärischer Einsatz von Kernwaffen mit seinen fatalen Folgen fast 30 Jahre nach Ende des Kalten Krieges wieder wahrscheinlicher geworden. Das nordkoreanische Regime konnte sein Kernwaffenprogramm trotz internationaler Sank-tionen erfolgreich vorantreiben, und auch der Iran forciert den Aufstieg zur Atommacht.
Die USA zeigen ebenfalls deutliche Anstrengungen für eine Erneuerung und Erweiterung ihres Nuklearwaffenarsenals [13].
Diese Beispiele zeigen, wie vielgestaltig und zum Teil umfänglich sich R/N Szenarien darstellen können, bei denen mit einer Strahlenexposition von zum Teil mehreren zehntausend Personen gerechnet werden muss.
Die akute Strahlenkrankheit
Insbesondere bei den R/N Szenarien Kraftwerkshavarie oder dem Einsatz einer IND ist bei betroffenen Personen tausendfach mit dem Auftreten der akuten Strahlenkrankheit (ASK) zu rechnen [14]. Oberhalb von Strahlendosen (homogene, nicht frak-tionierte Ganzkörperexposition über einen kurzen Zeitraum) von etwa 2 Gy ist das Auftreten der akuten hämatologischen Strahlenkrankheit zu erwarten.
Grundsätzlich gibt es drei Möglichkeiten, um die Wahrscheinlichkeit des Auftretens einer ASK abzuschätzen:
- die Nutzung von klinischen Zeichen und Symptomen,
- die Erhebung physikalischer Messdaten/Dosimetrie und
- der Nachweis von biologischen Veränderungen, z. B. Veränderungen im Zellkern.
Für die Anwendung physikalischer Messdaten ist entweder die Dosisrekonstruktion oder die Nutzung von Dosimetern notwendig. Im Falle eines unerwarteten Ereignisses wie einem Terroranschlag ist allerdings nicht davon auszugehen, dass Personen mit Dosimetern ausgestattet sein werden.
Die Nutzung von klinischen Zeichen und Symptomen, etwa die Frequenz und der Beginn von Erbrechen und Durchfall, könnten insbesondere für die schnelle radiobiologische Triage von großen Personengruppen sinnvoll sein, auch wenn sie nur einen ersten groben Anhalt liefern können [16, 17] und als nicht-strahlenspezifisch betrachtet werden müssen.
Als Goldstandard für die biologische Dosimetrie hat sich die dizentrische Chromosomenanalyse (DCA) etabliert [18]. Sie weist eine hohe Spezifität und Sensitivität auf, bleibt viele Monate nach einer Bestrahlung positiv, ist in der Lage, auch niedrige Dosen ab 100 mGy nachzuweisen und ist in vielen Labors weltweit harmonisiert. Große Nachteile des Verfahrens sind Aufwand und Dauer der Analyse, die mehrere Tage beträgt [19]. Die Methode ist für eine schnelle Triage somit, trotz einer guten Vernetzung der Speziallabore, eher ungeeignet [20].
Bei R/N Szenarien mit sehr hohen Patientenzahlen wird ein möglichst frühes und zielgerichtetes medizinisches Management mit dem Fokus auf klinisch relevante Fragestellungen notwendig sein, um das bestmögliche Behandlungsergebnis für viele Betroffene erreichen zu können [10, 21]. Diese klinisch relevanten Aspekte umfassen die Frage nach einer Hospitalisierung, der Notwendigkeit einer intensivmedizinischen Therapie und nach der Einleitung von medizinischen Gegenmaßnahmen [22]. Diese Punkte hängen maßgeblich von der frühen Abschätzung des zu erwartenden HASK-Schweregrads ab.
Molekularbiologische Verfahren haben das Potenzial, diese Effektabschätzung früh und in einem hohen Durchsatz vornehmen zu können [19]. Genexpressionsanalysen beispielsweise lassen sich – von der technologischen Seite her gesehen – einfach automatisieren und parallelisiert durchführen. Zahlreiche Forschungsgruppen konnten in den letzten beiden Dekaden bereits potenzielle, genexpressionsbasierte Biomarker identifizieren [23-30].
Zielsetzung
Diese Arbeit fasst die Ergebnisse jahrelanger kontinuierlicher Untersuchungen, beginnend mit Tierexperimenten[2], der Validierung im klinischen Modell bis hin zur Demonstration einer darauf aufbauenden Früh- und Hochdurchsatzdiagnostik zusammen. Sie thematisiert eine entscheidende wehrmedizinische Fähigkeitslücke im Bereich des medizinischen A-Schutzes. Es werden sehr vielversprechende Ansätze bis hin zur „Point-of-Care“ Diagnostik.
Material und Methoden
Identifizierung von Kandidatengenen
Tiere und Bestrahlung
Durch das Institut de Recherche Biomédicale des Armées (IRBA) in Frankreich wurden 18 Paviane (Papio anubis) verschiedenen Mustern einer Ganzkörper- (Total Body Irradiation, „TBI“) und Teilkörperbestrahlung (Partial Body Irradiation, „PBI“) (Äquivalent-Ganzkörperdosis zwischen 2,5 und 5 Gy) ausgesetzt. Vier Tiere erhielten eine TBI und 16 Tiere eine PBI (je zwei mit dem gleichen Muster). Für die Bestrahlung wurde eine Cobalt-60 Quelle genutzt [31].
Die Experimente wurden durch die Ethikkommission der französischen Armee geprüft und gebilligt (Nummer 2010/12.0). Die Tiere wurden gemäß Europäischer Gesetzgebung in Bezug auf Tierpflege und Tierschutz behandelt. Durch einen Tierarzt wurden supportive Maßnahmen zur Minimierung von Angst, Schmerz und Stress sichergestellt. Im Rahmen einer NATO Forschungsgruppe stellten die Franzosen ihr Modell vor und boten uns die Beteiligung an den laufenden Experimenten an.
Blutentnahme und Bestimmung des akuten hämatologischen Strahlensyndroms
Veränderungen im Differentialblutbild an bis zu 22 Zeitpunkten zwischen Tag 7 und 203 nach Bestrahlung wurden genutzt, um den HASK Schweregrad zu definieren [32]. Grundlage hierfür waren die METREPOL[3] Kriterien (H0: nicht-exponiert; H1-4: niedrig bis tödlich bestrahlt) [14]. Die Blutentnahmen (PAXgene Blood RNA Tubes, PreAnalytiX GmbH, Hombrechtikon, Schweiz) für die Genexpressionsanalysen erfolgten an Tag 0 (vor Bestrahlung, H0) sowie an Tag 1 und 2 nach Bestrahlung.
RNA Isolation und Qualitätskontrolle
2,5 ml Vollblut in PAXgene Röhrchen wurden im IRBA gemäß Anleitung des Herstellers entnommen, bei -20 °C gelagert und im Anschluss gefroren zur weiteren Bearbeitung an das Institut für Radiologie der Bundeswehr (InstRadBioBw) versendet. Die Isolation der Gesamt-RNA (mRNA und smallRNAs) erfolgte über die Kombination einer Phenol-Chloroform-Extraktion und dem mirVana™ Total RNA Isolation Kit (Thermo Fischer Diagnostics, Darmstadt, Deutschland). Während der Isolation wurde ein DNA-Verdau durchgeführt (RNAse free DNAse Set; Qiagen, Hilden, Deutschland). Die Qualität und Quantität wurden spektralphotometrisch überprüft (NanoDrop; PeqLab Biotechnologie, Erlangen, Deutschland). Die RNA-Integrität (RNA Integrity Number, RIN) wurde mittels Agilent 2100 Bioanalyzer (Life Science Group, Penzberg, Germany) gemessen und eine DNA Kontamination durch eine konventionelle PCR mit beta-Aktin Primern ausgeschlossen. RNA Proben mit einem A260/A280 Verhältnis von ≥ 2,0 (NanoDrop) und einer RIN ≥ 7,5 wurden für das Whole Genome Microarray Screening (IMGM® Laboratories, Martinsried, Germany) und mindestens mit RIN ≥ 7,3 für die qRT-PCR verwendet [32].
Schritt 1: Ganzgenomanalyse mittels Mikroarray
Anhand von 23 RNA Proben (H0, n = 5; H1-2, n = 2 x 4 an Tag 1 und 2 nach Bestrahlung; H2-3, n = 2 x 5, an Tag 1 und 2 nach Bestrahlung) wurde eine Ganzgenomanalyse (Whole Genome Microarray Screening) zur Untersuchung der proteinkodierenden mRNAs durchgeführt. Als Plattform wurde das Agilent Oligo Microarray GE 8 x 60 K (Agilent Technologies, Waldbronn, Deutschland) System in Kombination mit der GeneSpring GX12 Software verwendet. Der nicht-parametrische Mann-Whitney-Test wurde für den Vergleich der Genexpression zwischen H1-2 und H2-3 mit der H0 Referenzgruppe verwendet. Für die Auswahl der Kandidatengene zur Validierung wurden Transkripte gewählt, die in 50 % der Einzelproben exprimiert waren sowie einen p-Wert von ≤0,05 und eine ≥2-fold differentielle Genexpression zwischen den untersuchten Gruppen aufwiesen [32].
Schritt 2: Validierung des Mikroarray Screenings mittels qRT-PCR
Die identifizierten Gene aus dem Schritt 1 wurden unabhängig (23 bislang nicht verwendete RNA Proben) und methodisch (35 RNA-Proben) mittels TaqMan™ Low-Density Array (Hochdurchsatz 384-Well qRT-PCR Plattform) validiert. Dazu wurde 1 µg RNA mittels High Capacity cDNA Reverse Transcription Kit in komplementäre DNA (cDNA) umgewandelt. Die Reaktion und das gleichzeitige Auslesen der Expressionsdaten erfolgte mittels eines Applied Biosystems 7900 qRT-PCR Geräts. Die Genexpression wurde gegen das Haushaltsgen 18S normalisiert. Ein Unterschied der normalisierten Genexpression um den Faktor ≥2 gegenüber der Referenzgruppe wurde als differentielle Genexpression definiert („Fold Change“). Alle Reagenzien und Geräte wurden von der Firma Life Technologies (Darmstadt) bezogen [32].
Bioinformatische Analyse
Gene aus dem Screening mit einem p-Wert ≤0,05 und einer ≥2-fach differenziellen Genexpression wurden mittels PANTHER (Protein ANalysis THrough Evolutionary Relation-ships), (v10; http://www.pantherdb.org/), auf Gemeinsamkeiten in der biologischen Funktion analysiert (Referenz Homo Sapiens GO Datenbank).
Die qRT-PCR Daten wurden zur Validierung der Mikroarraydaten verwendet. Die Genexpressionsmessungen wurden auf ihre Assoziation mit klinisch relevanten Kategorien mittels logistischer Regressionsanalyse wie folgt untersucht:
- kein HASK (H0) gegen alle Formen des HASK (H1-3),
- kein HASK (H0) gegen leichtes HASK (H1-2) und
- kein HASK gegen schweres HASK (H2-3).
Neben deskriptiver Statistik wurden Odds-Ratios, 95 % -Konfidenz-Intervalle (CI) sowie korrespondierende p-Werte (Wald-Test) berechnet. Zur Bestimmung der diagnostischen Genauigkeit berechneten wir die Fläche unter der Receiver--Operating--Characteristic (ROC) Kurve. Alle Berechnungen erfolgten mit SAS Version 9.2 (SAS Institute, Cary, NC).
Validierung im Menschen und Übertragung auf einen ex vivo in vitro Hintergrund
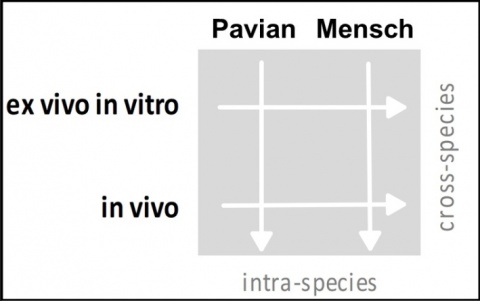
Um die Übertragbarkeit der Ergebnisse vom Pavian auf den Menschen zu prüfen, wurden in vivo Vollblutproben von Patienten, die eine diagnostische oder therapeutische Strahlenexposition erhielten, untersucht (Abbildung 3). Dabei wurden je fünf Patienten nach einer Computertomographie-Diagnostik (Dosisbereich 0,004-0,018 Sv), fünf Patienten nach einer Teilkörperbestrahlung auf Grund eines Prostatakarzinoms (Dosisbereich 0,25-0,3 Sv), sowie fünf Patienten nach einer Ganzkörperbestrahlung auf Grund einer Leukämie (Dosisbereich 2 × 1,5 oder 2 × 2 Sv) betrachtet. Es wurde die differentielle Genexpression von sechs, zuvor im Pavian-Modell identifizierten (WNT3, POU2AF1, CCR7, ARG2, CD177, WLS) sowie drei weiteren, häufig genutzten strahlensensiblen Genen (FDXR, PCNA, DDB2) untersucht. Weiterhin wurde die Übertragbarkeit der in vivo Genexpressionsergebnisse auf ein ex vivo in vitro Modell sowohl beim Pavian wie auch beim Menschen mittels ex vivo bestrahlter Blutproben (EDTA-Blut, n = 5 Pavian und n = 6 Mensch) von gesunden Individuen überprüft (Abbildung 3). Methodisch kam die qRT-PCR zur Anwendung [33].
Definition der Genexpressionsniveaus in einem Normalkollektiv
Umsetzung als Früh- und Hochdurchsatzdiagnostikum
Aktuell wird die Umsetzbarkeit der zuvor identifizierten und analysierten Gene in einem Früh- und Hochdurchsatzsetting untersucht. Hierzu wurde ex vivo in vitro entnommenes Blut bestrahlt (0 bis zu 5 Gy), verblindet und in 1 000 Einzelproben aliquotiert. Ziel ist es, als „proof of principle“ die parallelisierte, robotische RNA Isolation (QIAsymphony, Qiagen, Hilden, Deutschland) und die anschließende Genexpressionsanalyse mittels einer Sequenzierung der nächsten Generation (Next Generation Sequencing, NGS) inklusive Ergebnisbewertung innerhalb von 24 Stunden durchzuführen (Abbildung 4). Als Endpunkt wurden klinisch relevante Fragestellungen definiert (Identifizierung von nicht-exponierten Proben gegenüber exponierten Proben; Separation leichte von schwerer HASK).
Ergebnisse
Initiale Studie
Abbildung 5 liefert einen Gesamtüberblick über das zweistufige experimentelle Setting der initialen Studie, die Verteilung der Proben auf die einzelnen Stadien sowie einen Überblick über die verwendeten Analyseverfahren und Ergebnisse [32].
HASK Schweregradeinteilung

Ergebnisse Schritt 1: Whole Genome Mikroarray Screening
Von insgesamt etwa 20 000 untersuchten Genen waren im Durchschnitt 46 % exprimiert. Für die H2-3 Gruppe zeigten sich 2 bis 3-mal mehr hoch- und herunterregulierte Gene an Tag 1 als für die H1-2 Gruppe (1 418 gegen 550 und 1 603 gegen 735). An Tag 2 konnte eine weitere Zunahme dieser Tendenz bei einer gleichzeitigen Abnahme der Gesamtzahl der differentiell-exprimierten Gene beobachtet werden (Abbildung 6). Bei einer nur geringen Überlappung zwischen H1-2 und H2-3 (91 hoch- und 169 herunterregulierte Gene) verblieben 692 hoch- und 174 herunterregulierte Kandidatengene für eine Differenzierung der beiden Gruppen voneinander. Mittels PANTHER konnte eine Verflechtung der Kandidatengene in immunologische Prozesse gezeigt werden [32].

Ergebnisse Schritt II: Validierung mittels qRT-PCR
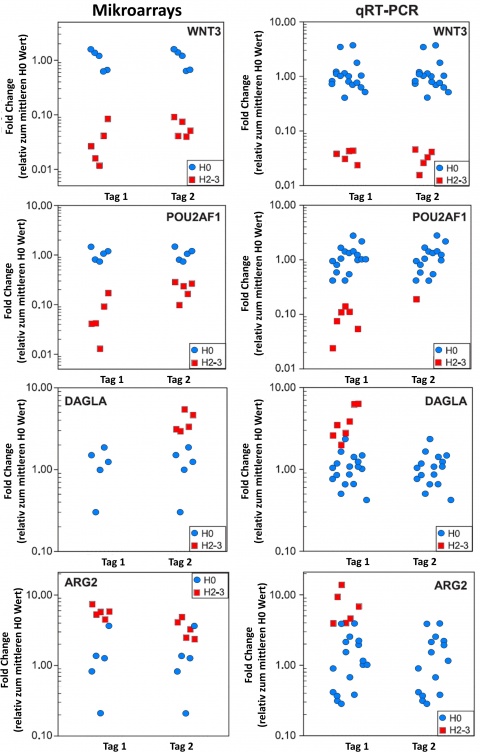
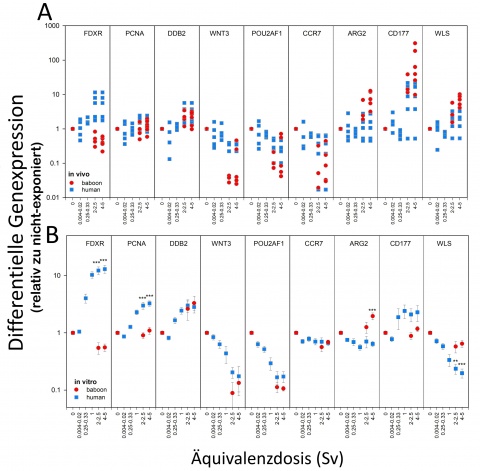
Von den 89 ausgewählten Genen zeigten 47 keine Amplifikation in der qRT-PCR; 13 Gene zeigten niedrigere oder weniger konstante „Fold Changes“ als im Mikroarray. Die verbliebenen 22 Gene konnten mittels qRT-PCR für eine Unterscheidung zwischen nicht-exponiert (H0) und exponiert (H1-3) bestätigt werden. Die analysierten „Fold Changes“ befanden sich überwiegend im Bereich 3-5, für einzelne Gene, aber auch zum Teil deutlich darüber hinaus (CD177; 30,7-fach hochreguliert). Als Maß für die diagnostische Genauigkeit zeigte sich bei drei Genen eine ROC-Fläche[4] von 1 als Zeichen einer kompletten Separation (mittlerer ROC Wert für alle 22 Gene = 0,93) [32].
Für die Diskriminierung einer schwereren Verlaufsform des HASK (H2-3) konnten 7 Gene gefunden werden, mit einer bis zu 30-fachen Deregulation (WNT3) (Abbildung 7). Eine komplette Separation ließ sich ebenfalls bei drei Genen zeigen (ROC 1; Mittlerer ROC Wert für alle 7 Gene = 0,93) [32].
Validierung im Menschen

Der Vergleich der in vivo Ergebnisse mit Messungen aus einem ex vivo in vitro Vergleich („intra-species“ Validierung) offenbarten bei Pavian und Mensch für FDXR, PCNA, DDB2, WNT3 und POU2AF1 eine Dosis-Wirkungs-Beziehung. Für CCR7, ARG2, CD177 und WLS fanden sich abweichende Expressionsmuster (Daten nicht gezeigt) [33].
Bestimmung des basalen Genexpressionsniveaus in einem Normalkollektiv
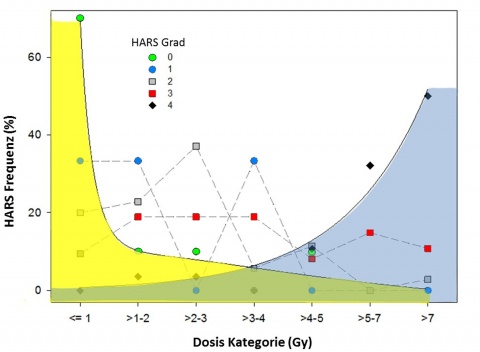
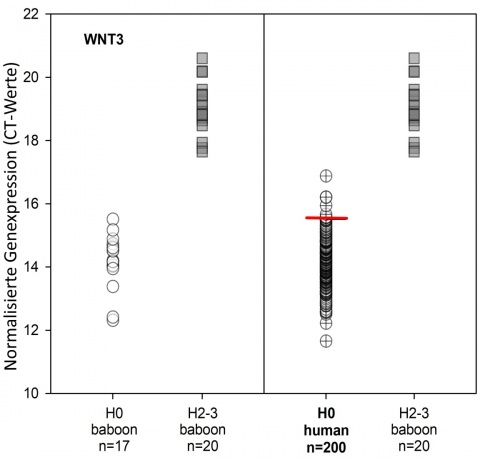
Anhand der 122 männlichen und 88 weiblichen Blutspender (Durchschnittsalter 46 Jahre) wurde die interindividuelle Varianz der Genexpression für DAGLA, WNT3, CD177, PLA2G16, WLS, POU2AF1, STAT4, PRF1, FDXR und DDB2 in einem gesunden Normalkollektiv bestimmt. Diese Werte wurden als – um den Faktor 10 größere H0 Gruppe – für einen Vergleich mit den klinischen HASK Gruppen aus dem vorangegangenen Experiment genutzt. Es zeigte sich trotz einer zum Teil hohen interindividuellen Varianz eine weiterhin sehr gute Diskriminierbarkeit mit zum Teil vollständiger Separation zwischen H0 und H2-3 (WNT3, Abb.9) [34].
Umsetzung als Früh- und Hochdurchsatzdiagnostik
Der in Abbildung 4 dargestellte experimentelle Ansatz zur Etablierung einer Früh- und Hochdurchsatzdiagnostik wird aktuell bearbeitet, um die Hochdurchsatzfähigkeit von Genexpressionsanalysen in einem kurzen Zeitraum zu erproben. Zum aktuellen Zeitpunkt wurde bereits die Probengenerierung finalisiert und die robotische Isolation von 1 008 Proben innerhalb von 12 Stunden durchgeführt. Ausstehend ist die Analyse mittels gezieltem NGS.
Diskussion
Die dargestellten Ergebnisse zeigen den aktuellen Stand der Entwicklung einer genexpressionsbasierten Früh- und Hochdurchsatzdiagnostik des HASK. Im Rahmen von R/N Großschadenslagen könnte diese neue Diagnostik eine zentrale Rolle für das medizinische Management von strahlenexponierten Personen darstellen. In einem mehrstufigen Konzept wurde auf Grundlage von tierexperimentellen Daten ein Genexpressionsmuster identifiziert [32] und mittels diagnostisch und/oder therapeutisch strahlenexponierter Patienten erfolgreich im Menschen validiert [33]. Gleichzeitig konnten die in vivo Ergebnisse auf ein ex vivo in vitro Zellkultur-Setting übertragen werden [33]. Zusätzlich wurde die interindividuelle Varianz der strahlensensiblen Kandidatengene in einem großen Normalkollektiv bestimmt (34). Aktuell wird die praktische Umsetzung in einem Früh- und Hochdurchsatzsetting anhand von 1 008 Blindproben getestet, die innerhalb von 24 Stunden isoliert und analysiert werden.
Bemerkenswert ist bei unserem Ansatz, dass es sich nicht um ein klassisches biodosimetrisches Konzept handelt. Durch zahlreiche Forschungsgruppen konnte bereits in der Vergangenheit gezeigt werden, dass es zu strahleninduzierten Genexpressionsveränderungen kommt [23-30]. In unserem Fall wird aber ein zur Biodosimetrie komplementärer Ansatz genutzt, der keine Abschätzung der Dosis, sondern der Krankheitseffekte verfolgt [15]. Es ergeben sich für die radiobiologische Diagnostik Vorteile, da durch eine Effektabschätzung über Genexpressionsänderungen Faktoren wie Strahlenqualität (Verhältnis Gamma- zu Neutronenstrahlung), Ganzkörper/Teilkörperbestrahlung, Fraktionierung, individuelle Radiosensitivität, Inkorporation, usw. direkt integriert werden.
Klinische Aspekte
Die Endpunkte unseres Ansatzes sind klinisch relevante Fragen, konkret etwa die Identifizierung der Personen, die keine Strahlendosis erhalten haben, sogenannte „worried wells“ (H0 nach METREPOL) und somit keinerlei weitere Behandlung benötigen. Weiter sind die zu identifizieren, bei denen sich eine leichte Verlaufsform des HASK entwickeln wird (H1-2) und diejenigen, bei denen eine schwere HASK Verlaufsform zu erwarten ist (H3-4). Mit diesem Ansatz lässt sich zum einen auf die psychologischen Belange der zahlreichen zu erwartenden „worried wells“ eingehen (frühe Diagnosestellung), und zum anderen lassen sich die begrenzten Krankenhausressourcen sinnvoll nutzen (stationäre Behandlungsoptionen und insbesondere Überwachungskapazitäten bei schweren HASK Verlaufsformen). Zusätzlich unterstützt diese frühe Entscheidungsfindung die zeitgerechte Applikation von Gegenmaßnahmen, wie z. B. eine Dekorporationstherapie nach Radionuklidinkorporation [36] oder die Gabe von Zytokinen [22], die möglichst zeitnah (innerhalb von 48 Stunden nach dem Ereignis) erfolgen sollten. Diesem zeitkritischen Aspekt wird der Goldstandard der Biodosimetrie, die dizentrische Chromosomenanalyse nicht gerecht, da sie als wesentlichen Schritt die mehrtägige Kultivierung von Lymphozyten beinhaltet [19].
Auch ein Hochdurchsatz mit mehreren 1 000 Patientenproben lässt sich trotz etablierter Biodosimetrie-Netzwerke aktuell nicht realisieren [20]. Bisherige Ringversuche mit Blindproben haben nur wenige Einzelproben (bis zu 10 Proben) untersucht [19, 37, 38]. Unser Ansatz zielt darauf ab, dies um den Faktor 100 zu steigern und gleichzeitig das Zeitfenster um den Faktor 4 zu reduzieren, um eine Großschadenslage realistischer nachzubilden.
Limitationen
Des Weiteren muss die Grunderkrankung der Patientinnen und Patienten betrachtet werden. Für WNT3 beispielsweise zeigt sich eine 57-fache Herunterregulation vor Bestrahlung in den Leukämiepatienten gegenüber dem Normalkollektiv [33, 34]. Dies erklärt die Überlegung, differenzielle Genexpression anstatt einfacher, normalisierter Genexpressionswerte zu betrachten.
Ausblick
Als ein weiterer Ausblick könnte eine genexpressionsbasierte qRT-PCR Diagnostik als „Point-of-Care“ Konzept (patientennahe Labordiagnostik) umgesetzt werden [39]. „Microfluidic“-Technologie ermöglicht eine chip-basierte Durchführung komplexer Laborverfahren in miniaturisierten Geräten. Dies könnte beispielsweise einen „forward deployable“ Einsatz in einem zivilen, katastrophenmedizinischen Kontext ermöglichen und die HASK-Diagnostik durch die vereinfachte Logistik noch zusätzlich beschleunigen. Weiterhin ist diese „Point-of--Care“-Diagnostik als eine konzeptionell sinnvolle Ergänzung zum Ansatz eines „Medical Radiological Incident Investigation Team“ (MED-RIIT) zu sehen, der gemäß NATO STANAG 2873[5] gefordert wird [40].
Zusammenfassend zeigt sich, dass Genexpression – trotz aller Limitierungen – zukünftig die Lücke in Bezug auf eine frühe und hochdurchsatzgeeignete Diagnostik des HASK schließen könnte. In weiterer Zukunft könnte sich hieraus sogar eine „Point-of-Care Diagnostik“ für den hochmobilen, zivilen und militärischen Einsatz von Genexpressionsanalysen zur HASK Diagnostik nach R/N-Szenarien entwickeln lassen.
Kernaussagen
- Militärische und zivile radiologische und nukleare Szenarien sind mögliche Großschadenslagen mit mehreren 1 000 potenziell strahlenexponierten Personen.
- Die aktuellen Diagnostika der hämatologischen akuten Strahlenkrankheit weisen Lücken bei der Früh- und Hochdurchsatznutzbarkeit auf.
- Mittels einer Gensignatur lässt sich die später auftretende hämatologische akute Strahlenkrankheit innerhalb der ersten zwei Tage nach einem Bestrahlungsereignis diagnostizieren.
- Mittels eines genexpressionsbasierten Verfahrens werden aktuell 1 000 bestrahlte Proben innerhalb nur eines Tages prozessiert und analysiert.
- Zukünftig könnte das neue Verfahren auch als „Point-of-Care“ Konzept für eine patientennahe Labordiagnostik genutzt werden.
Literatur
- Gale RP, Armitage JO: Are We Prepared for Nuclear Terrorism? N Engl J Med 2018; 378(13): 1246-1254.
- The 2016 Nuclear Security Summit. (last accessed on 21 November 2018).
- Hérodin F (Chair) et al.: Biological Effects of Ionising Radiation and Countermeasures. NATO-STO-HFM Technical Report 222, 2018.
- Kibaroğlu M, Voica D-R: Response to Nuclear and Radiological Terrorism. Amsterdam: IOS Press, 2011.
- Harrison J, Fell T, Leggett R, Lloyd D, Puncher M, Youngman M: The polonium-210 poisoning of Mr Alexander Litvinenko. J Radiol Prot 2017; 37(1): 266-278.
- US Department of Health & Human Services: Radiological Exposure Devices (REDs) – Radiation Emergency Medical Management. (last accessed on 21 November 2018).
- INTERNATIONAL ATOMIC ENERGY AGENCY: The Radiological Accident in Goiânia. Vienna: INTERNATIONAL ATOMIC ENERGY AGENCY; 1988.
- SPIEGEL ONLINE: Angriff auf Irans Atomprogramm: Stuxnet-Virus könnte tausend Uran-Zentrifugen zerstört haben. (last accessed on 21 November 2018).
- Federal Emergenca Planning Agency: Planning Guidance for a Response to a Nuclear Detonation. (last accessed on 21 November 2018).
- Coleman CN, Knebel AR, Hick JL et al. Scarce resources for nuclear detonation: project overview and challenges. Disaster Med Public Health Prep 2011;5 Suppl 1(S1):S13-S199.
- Wellerstein A: NUKEMAP. (last accessed 21 November 2018).
- IAEA: Incident and Trafficking Database (ITDB). (last accessed 21 November 2018).
- US Department of Defenxe: 2018 Nuclear Posture Review (last accessed 21 November 2018).
- Friesecke I, Beyrer K, Fliedner TM: How to cope with radiation accidents: The medical management. Br J Radiol 2001; 74(878):121-122.
- Port M, Pieper B, Dörr HD, Hübsch A, Majewski M, Abend M: Correlation of Radiation Dose Estimates by DIC with the METREPOL Hematological Classes of Disease Severity. Radiat Res 2018; 189(5): 449-455.
- Goans RE, Waselenko JK: Medical management of radiological casualties. Health Phys 2005; 89(5): 505-512.
- Port M, Pieper B, Knie T et al.: Rapid Prediction of Hematologic Acute Radiation Syndrome in Radiation Injury Patients Using Peripheral Blood Cell Counts. Radiat Res 2017; 188(2): 156-168.
- International Atomic Energy Agency. Cytogenetic analysis for radiation dose assessment : A manual. IAEA 2001 < https://www.pub.iaea.org/MTCD/Publications/PDF/TRS405_scr.pdfInternational Atomic Energy Agency> (last accessed 21 November 2018).
- Rothkamm K, Beinke C, Romm H et al.: Comparison of Established and Emerging Biodosimetry Assays. Radiat Res. 2013; 180(2): 111-119.
- Kulka U, Ainsbury L, Atkinson M et al.: Realising the European Network of Biodosimetry (RENEB). Radiat Prot Dosimetry 2012; 151(4): 621-625.
- Dainiak N: Medical management of acute radiation syndrome and associated infections in a high-casualty incident. J Radiat Res. Oxford University Press 2018; 59(suppl_2): ii54-ii64.
- Farese AM, MacVittie TJ: Filgrastim for the treatment of hematopoietic acute radiation syndrome. Drugs Today (Barc) 2015; 51(9): 537-548.
- Liu T-P, Hsu Y-Y, Lai L-C, Tsai M-H, Chuang EY: Identification of gene expression biomarkers for predicting radiation exposure. Sci Rep 2014; 4(1): 6293.
- Paul S, Amundson SA: Development of gene expression signatures for practical radiation biodosimetry. Int J Radiat Oncol Biol Phys 2008; 71(4): 1236-1244.
- Dressman HK, Muramoto GG, Chao NJ et al.: Gene Expression Signatures That Predict Radiation Exposure in Mice and Humans. Kemp Ch, editor. PLoS Med 2007; 4(4): e106.
- Amundson SA, Do KT, Shahab S et al.: Identification of potential mRNA biomarkers in peripheral blood lymphocytes for human exposure to ionizing radiation. Radiat Res 2000; 154(3): 342-346.
- Berglund SR, Rocke DM, Dai J et al.: Transient genome-wide transcriptional response to low-dose ionizing radiation in vivo in humans. Int J Radiat Oncol Biol Phys 2008; 70(1): 229-234.
- Manning G, Kabacik S, Finnon P, Bouffler S, Badie C: High and low dose responses of transcriptional biomarkers in ex vivo X-irradiated human blood. Int J Radiat Biol 2013; 89(7): 512-522.
- Brzóska K, Kruszewski M: Toward the development of transcriptional biodosimetry for the identification of irradiated individuals and assessment of absorbed radiation dose. Radiat Environ Biophys 2015; 54(3): 353-363.
- Lacombe J, Sima C, Amundson SA, Zenhausern F: Candidate gene biodosimetry markers of exposure to external ionizing radiation in human blood: A systematic review (Editor: Amendola R). PLoS One 2018; 13(6): e0198851.
- Valente M, Denis J, Grenier N et al.: Revisiting Biomarkers of Total-Body and Partial-Body Exposure in a Baboon Model of Irradiation (Editor: Fornace AJ). PLoS One 2015; 10(7): e0132194.
- Port M, Herodin F, Valente M et al.: First Generation Gene Expression Signature for Early Prediction of Late Occurring Hematological Acute Radiation Syndrome in Baboons. Radiat Res 2016; 186(1): 39-54.
- Port M, Majewski M, Herodin F et al.: Validating Baboon Ex Vivo and In Vivo Radiation-Related Gene Expression with Corresponding Human Data. Radiat Res. The Radiation Research Society 2018; 189(4): 389-398.
- Agbenyegah S, Abend M, Atkinson MJ et al.: Impact of Inter-Individual Variance in the Expression of a Radiation-Responsive Gene Panel Used for Triage. Radiat Res 2018; 190: 226-235.
- Gomes I, Sharma TT, Mahmud N et al.: Highly abundant genes in the transcriptosome of human and baboon CD34 antigen-positive bone marrow cells. Blood 2001; 98(1): 93-99.
- Rump A, Stricklin D, Lamkowski A, Eder S, Abend M, Port M: The Incorporation of Radionuclides After Wounding by a “Dirty Bomb”: The Impact of Time for Decorporation Efficacy and a Model for Cases of Disseminated Fragmentation Wounds. Adv Wound Care 2017; 6(1): 1-9.
- Ainsbury E, Badie C, Barnard S et al.: Integration of new biological and physical retrospective dosimetry methods into EU emergency response plans – joint RENEB and EURADOS inter-laboratory comparisons. Int J Radiat Biol 2017; 93(1): 99-109.
- Abend M, Badie C, Quintens R et al.: Examining Radiation-Induced In Vivo and In Vitro Gene Expression Changes of the Peripheral Blood in Different Laboratories for Biodosimetry Purposes: First RENEB Gene Expression Study. Radiat Res 2016; 185(2): 109-123.
- Pandey CM, Augustine S, Kumar S et al.: Microfluidics Based Point-of-Care Diagnostics. Biotechnol J 2018; 13(1): Epub 2017 (doi: 10.1002/biot.201700047).
- NATO-STANAG 2873: COMMANDER’S GUIDE TO MEDICAL OPERATIONS IN SUPPORT OF CBRN DEFENSIVE OPERATIONS. NATO, 2018.
Danksagung
Die vorliegende Arbeit ist das Ergebnis einer jahrelangen, fruchtbaren Kooperation des Instituts für Radiobiologie der Bundeswehr mit zahlreichen militärischen und zivilen Partnern. Ohne sie wäre diese Arbeit nicht möglich gewesen.
Mein besonderer Dank gilt meinem Arbeitsgruppenleiter, Oberstarzt Prof. Dr. Michael Abend, der diese Arbeit mit seiner außerordentlichen konzeptionellen und fachlichen Kompetenz und seiner Vernetzung innerhalb der internationalen radiobiologischen Gemeinschaft erst ermöglicht und allzeit maßgeblich mitgestaltet hat.
Zitierweise:
Majewski M: Etablierung einer genexpressionsbasierten Früh- und Hochdurchsatzdiagnostik der hämatologischen akuten Strahlenkrankheit. Wehrmedizinsche Monatsschrift 2019; 63(1): 2-10.
Verfasser
Oberstabsarzt Dr. Matthäus Majewski
Institut für Radiobiologie der Bundeswehr
Neuherbergstr. 11, 80937 München
E-Mail: [email protected]
[1] Votum der zuständigen Ethik-Kommission: Siehe zweiter Absatz des Kapitels Material und Methoden.
[2] Votum der zuständigen Ethik-Kommission: Siehe zweiter Absatz des Kapitels Material und Methoden.
[3] METREPOL steht für Medical Treatment Protocols for Radiation Accident und wurde 2005 international als Basis für das medizinische Management Betroffener bei Strahlenunfällen definiert. In die Vorhersage des zu erwartenden Schweregrades einer Strahlenkrankheit gehen unter anderem hämatologische, neurovasculäre, cutane und gastrointestinale Symptome zu einem frühen Zeitpunkt nach Exposition ein (siehe auch [14]).
[4] ROC (Receiver Operating Characteristic) ist ein statistisches Verfahren, mit dem die diagnostische Qualität von Messverfahren mit binärem Ausgang (z. B. Laborparametern) bestimmt werden kann. Es werden die Sensitivität (Y-Achse) und die falsch positive Rate (X-Achse) dargestellt. Die Fläche unter der Kurve macht dabei eine Aussage über die diagnostische Genauigkeit des Verfahrens. Ein idealer Test ohne Fehlentscheidungen ergibt eine Fläche unter der Kurve von 1; ein rein auf Zufall basierendes Ergebnis würde zu einem Wert von 0,5 führen.
[5] STANAG = Standardization Agreement: In der NATO vereinbarter Standard für Verfahren, Methoden und Ausrüstung, der die Kompatibilität, insbesondere bei multinationalen Operationen, sicherstellen soll.
Datum: 22.02.2019