Diagnostik 2.0: Next Generation Sequencing als moderne diagnostische Erweiterung im medizinischen B-Schutz
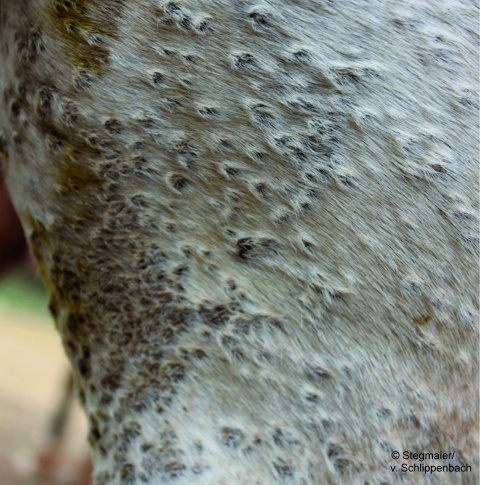
Am Beispiel von pockenartigen Läsionen bei einem Pferd
Rosina Ehmannª, Kristin Brandes ᵇ, Markus Antwerpenª, Mathias Walterª, Katja von Schlippenbach ͨ , Elisabeth Stegmaier ͩ, Sandra Eßbauerª, Joachim Bugertª, Jens Peter Teifke ͤ, Hermann Meyer ª
ª Institut für Mikrobiologie der Bundeswehr, München
ᵇ Tierpathologie Augsburg, Deutschland
ͨ Tierarztpraxis Zusamaltheim, Deutschland
ͩ Makoa Farm, Tansania
ͤ Friedrich-Loeffler-Institut, Greifswald – Insel Riems, Deutschland
Einleitung

Seit wenigen Jahren bietet Next Generation Sequencing (NGS), ein Sammelbegriff für verschiedene Techniken der Hochdurchsatz-Sequenzierung, enorme Chancen zur breit angelegten Erregerdiagnostik. Allerdings ist NGS sowohl in Bezug auf technisches Know-how (labortechnisch, molekularbiologisch und bioinformatisch) als auch im Hinblick auf die Geräteausstattung eine herausfordernde Technik. Zur Implementierung müssen für verschiedene Probentypen (Serum, Stuhl, Nasenabstrich, Umweltprobe, …) zusätzlich jeweils individuelle Aufarbeitungsverfahren getestet und etabliert werden. Zur Evaluierung der Anwendungsmöglichkeiten von NGS in der Pockenvirusdiagnostik wurden im Rahmen dieser Arbeit beispielhaft Hautveränderungen eines Pferdes diagnostisch aufgearbeitet und ausgewertet. Die eingesetzten Methoden und Geräteplattformen wurden im Hinblick auf ihre Anwendbarkeit in der stationären und mobilen Diagnostik ausgewählt und verglichen. Der als ursächliches Agens identifizierte Erreger erwies sich als Vertreter einer neuen Virusspezies und wurde weitergehend charakterisiert.
Hier geht es zum vollständigen Artikel.
Datum: 17.12.2019
Wehrmedizinische Monatsschrift 12/2019