SEPSISDIAGNOSTIK IN DEN TROPEN
Diagnosis of sepsis in the tropics*
Aus dem Fachbereich Tropenmedizin am Bernhard-Nocht-Institut für Tropenmedizin (Fachbereichsleiter: Oberfeldarzt Dr. H. Sudeck), Bundeswehrkrankenhaus Hamburg (Chefarzt: Generalarzt Dr. J. Hoitz)
Hagen Frickmann
WMM, 57. Jahrgang (Ausgabe 11/2013: S. 309-313)
Zusammenfassung
Hintergrund: Kulturelle Sepsisdiagnostik im tropischen Einsatz ist durch methodische Probleme, insbesondere lange Lagerung- und Transportzeiten sowie klimatische Einflüsse, in ihrer Sensitivität limitiert. Molekulare Diagnostik könnte dieses Problem reduzieren.
Methoden: Zwei real-time 16S rRNA-Gen-PCR-Protokolle mit anschließender Sequenzierung wurden bei 1 500 kulturell charakterisierten Blutkulturrückstellproben aus dem tropischen Ghana zur molekularen Erregeridentifikation eingesetzt.
Ergebnisse: In mehr als zwei Dritteln der kulturell nachgewiesenen Erreger bestand Konkordanz zwischen dem biochemischen Differenzierungsergebnis und der sequenzbasierten Erregeridentifizierung auf Genus- oder Speziesebene. Durch die PCR und Sequenzierung konnte die Zahl der potenziell ätiologisch relevanten Erregernachweise in Ergänzung zur Kultur um zwei Drittel erhöht werden, jedoch stieg auch die Zahl der Kontaminationsnachweise überproportional an.
Schlussfolgerungen: Die sequenzbasierte Blutkulturdiagnostik ist eine sinnvolle Ergänzung zum kulturellen Erregernachweis unter präanalytisch schwierigen tropischen Bedingungen. Die kontaminationsfreie Nukleinsäureaufreinigung bedarf weiterer Optimierung. Ferner bleibt die Markteinführung mobiler Sequenzierautomaten hinreichender Leistungsfähigkeit abzuwarten.
Schlagwörter: Blutkulturdiagnostik; PCR; Sequenzierung; Tropen; Sepsis.
Summary
Background: Sensitivity of culture-based blood-culture diagnostics is limited by methodical problems, in particular increased storage and transport time and climatic influences, during tropical deployments. Molecular diagnostics might reduce this problem.
Methods: Two real-time 16S rRNA gene PCR protocols with subsequent sequencing were applied to 1,500 blood culture residual samples (characterized by culture) from tropical Ghana to allow for a molecular identification of blood culture pathogens.
Results: Concordance of biochemical identification results and sequence-based identifications on genus or species level could be achieved in more than two thirds of culture-positive samples. PCR and sequencing in addition to culture led to an increase of the number of potentially etiologically relevant pathogen detections by two thirds. However, there was a disproportional increase of detected contaminants as well.
Conclusions: Sequence-based blood culture diagnostics are suitable additions to cultural pathogen detection under difficult pre-analytic conditions in the tropics. Contamination-free nucleic acid enrichment requires further optimization. Commercial availability of mobile sequencers with sufficient performance has to be awaited.
Keywords: Blood culture diagnostics; PCR; sequencing; tropics; sepsis.
Einführung
„Und ich sah, und siehe, ein fahles Pferd. Und der darauf saß, dessen Name war: Der Tod […]. Und ihnen wurde Macht gegeben […] zu töten mit […] Pestilenz […].“
Offenb. 6;8
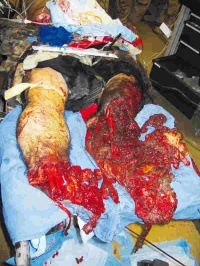
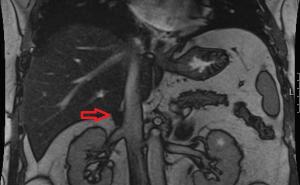
Die Auslandseinsätze der Bundeswehr werden zunehmend robuster, was die Beteiligung an Kampfhandlungen in Krisenlagen mittlerer und hoher Intensität beinhaltet. Dies geht einher mit schweren Traumata und komplexen Verletzungsmustern durch Ansprengungen (Abb. 1) sowie Schuss- oder Splitterverletzungen.
Die dabei entstehenden, teils großflächig offenen Verletzungen müssen als primär mikrobiell kontaminiert angesehen werden. Zudem ist eine Wundversorgung im Feld unter aseptischen Kautelen nicht möglich. Unter solchen Bedingungen können sich lokale Wundinfektionen auch bei gesunden jungen Soldaten sehr schnell zur Bakteriämie und Sepsis weiterentwickeln.
Nach Angaben des deutschen Kompetenznetzes Sepsis (SepNet; www.kompetenznetz-sepsis.de/) verläuft zumindest die schwere Sepsis selbst im Heimatland für jeden zweiten Betroffenen tödlich. In Deutschland mag für einen Teil der häufig alten, multimorbiden Sepsispatienten die Bakteriämie aufgrund zusammenbrechender Blut-Gewebeschranken die Endstrecke der Agoniephase bei schwerer, infauster Grunderkrankung darstellen, sodass aus ethischen Erwägungen und im Konsens mit Patienten und Angehörigen auf eine Maximaltherapie verzichtet wird. Im Einsatz dagegen stellt die Sepsis grundsätzlich immer einen akut behandlungsbedürftigen Notfall dar.
Erschwert wird die kalkulierte antimikrobielle Therapie der Sepsis in den Einsatzgebieten durch teils erhebliche Anteile von Erregern mit atypischer Resistenzsituation oder Multiresistenz [1 – 3]. Dies ist insofern problematisch, dass zumindest beim Patienten im septischen Schock (also mit katecholaminpflichtiger Sepsis) mit jeder Stunde Verzögerung einer wirksamen antibiotischen Therapie die Sterblichkeit um 7 % steigt [4]. Eine – zum Beispiel aufgrund atypischer Resistenzsituation des Sepsiserregers – insuffiziente Antibiotikatherapie hilft dabei genauso wenig wie gar keine. Angesichts der raschen progredienten Dynamik der Sepsis kommt das Ergebnis der mikrobiologischen Diagnostik derzeit und für die nächste Zukunft für die Anleitung zur spezifischen Antibiotikatherapie zu spät, sodass es momentan keine Alternative zur initialen kalkulierten Gabe von Breitspektrumantibiotika-Kombinationen [5] mit dem assoziierten Risiko der Resistenzselektion gibt.
Grundsätzlich setzt die zielgerichtete antimikrobielle Therapie die Kenntnis des Infektionserregers voraus, womit einer zügigen Erregeridentifizierung ein besonderer Stellenwert zukommt. Dazu wurde eine Vielzahl molekularer Verfahren zum nicht-kulturellen Erregerdirektnachtweis entwickelt, von denen die PCR in der mikrobiologischen Routinediagnostik die breiteste Anwendung gefunden hat. Während überzeugend demonstriert werden konnte, dass die PCR in der Routineanwendung zur Reduktion des Antibiotika-Gebrauchs führen kann [6], blieb das Verfahren den Nachweis einer Kostenreduktion im Gesundheitswesen bisher schuldig [7]. Allerdings konnte eine Schweizer Studie den unmittelbaren Einfluss der Schnelldifferenzierung von Erregern aus Blutkulturmaterial auf die kalkulierte Antibiotikatherapie zumindest für den Nachweis von gramnegativen Stäbchen belegen, wodurch überwiegend eine kalkulierte Erweiterung des Wirkungsspektrums mit potenziellem Benefit für die Therapie des Patienten erreicht werden konnte [8]. Aufgrund der zunehmend implementierten Resistenzsurveillance in den Auslandseinsätzen [3] kann bereits die frühzeitige Kenntnis der Erregergattung oder -spezies zur Optimierung der kalkulierten Antibiotikatherapie im Einsatz beitragen.
Die kulturelle Blutkulturdiagnostik im Einsatz wird durch ungünstige präanalytische Bedingungen erschwert. Die Qualitätsstandards in der mikrobiologisch-infektiologischen Diagnostik (MiQ), leitlinienähnliche diagnostische Handlungsanweisungen, empfehlen eine Transportzeit beimpfter Blutkulturmedien von nicht mehr als zwei bis vier Stunden. Die maximale Lagerungszeit wird mit 16 Stunden angegeben. Jenseits dieses Zeitfensters muss mit Sensitivitätsverlusten durch das Absterben empfindlicher Keime oder – zumindest bei automatischen Inkubationssystemen – einer eingeschränkten Auslesbarkeit gerechnet werden [9].
Da eine kulturelle Sepsisdiagnostik jedoch regelhaft erst ab ‚Role 3‘-Feldlazaretten vorgehalten wird, sind Transportzeiten von 16 Stunden für periphere Sanitätseinrichtungen kaum realisierbar, was zum Verzicht auf die Blutkulturdiagnostik oder zur Frustration über negativ bleibende Einsendungen führt. Hinzu kommt, dass die Empfehlungen der MiQ auf Studien aus Ländern der gemäßigten Klimazone beruhen. In subtropischen oder tropischen Einsatzgebieten, wie zum Beispiel Afghanistan oder Mali, herrschen dagegen extreme Klimabedingungen vor, deren konkrete Auswirkungen auf die Sensitivität kultureller Blutkulturdiagnostik nicht abschließend untersucht sind. Eine eigene Studie mittels real-time-PCR auf enteroinvasive bakterielle Erreger an Blutkulturmaterialien aus dem tropischen Ghana zeigte für Salmonellen, die häufigsten Sepsiserreger in Zentralafrika und Südostasien [10, 11], einen diagnostischen Zugewinn von etwa einem Drittel im Vergleich zur rein kulturellen Diagnostik [12].
In der im Folgenden beschriebenen Studie erfolgte die molekulare Diagnostik aus tropischen Blutkulturproben mit panbakteriellen PCR-Verfahren und anschließender Sequenzierung zwecks Erregeridentifikation auf Genus- bis Speziesebene. Zielregion war das 16S rRNA-Gen, eine phylogenetisch hochkonservierte Region des prokaryotischen Genoms, die für einen Teil der kleinen Untereinheit des bakteriellen Ribosoms kodiert.
Methoden
Untersucht wurden 1 500 Rückstellproben vorinkubierter Blutkulturproben aus dem tropischen Ghana. Bei einem Probentransport vom Krankenhaus zum Labor über 80 km und Transportzeiten zwischen 12 und 72 Stunden waren die präanalytischen Bedingungen ähnlich ungünstig wie im Einsatz. Nach Probeneingang im ghanaischen Labor erfolgte eine automatisierte Inkubation (BACTEC 9050, BD, Heidelberg) mit anschließender biochemischer Erregercharakterisierung mit mikrobiologischen Standardverfahren. Daraus resultierten 191 kulturell positive Befunde. In 98 Fällen wurden mutmaßlich ätiologisch relevante Sepsiserreger (zum Beispiel Staphylococcus aureus, Enterobakterien, Nonfermenter etc.) und in 93 Fällen klassische Hautflora-Kontaminanten (unter anderem Koagulase-negative Staphylokokken) nachgewiesen, die oft bei unsteriler Probenentnahmetechnik auftreten.
Die Anreicherung potenziell vorhandener Erreger in den Rückstellproben und Abtrennung inhibitorischer Erythrozytenbestandteile erfolgten durch ein einfaches, kostengünstiges Zentrifugationsverfahren [12] vor Durchführung der Molekulardiagnostik. Vorherige Versuche mit traditionellen DNA-Aufreinigungssystemen (Säulentechnik und automatisiert mittels QiaSymphony®, beides Qiagen, Hamburg) waren aufgrund vollständiger Probeninhibition verworfen worden (Daten nicht gezeigt). Die PCR-Diagnostik erfolgte nach einem Stufenschema, da panbakterielle PCR-Verfahren mit langen Zielstrukturen mittels Sequenzierung zwar eine zuverlässigere Diskriminierung erlauben, PCR-Verfahren mit kurzen Zielstrukturen dafür eine höhere Sensitivität aufweisen [13]. Zunächst wurde eine mittelgradig sensitive SYBR Green real-time 16S rRNA-Gen-PCR [14, 15] eingesetzt, die ein 917-Basenpaarfragment amplifizierte. Alle negativ gebliebenen Proben wurden mit einer auf hohe Sensitivität optimierten SYBR Green real-time 16S rRNA-Gen-PCR [16] mit einem 217-Basenpaarfragment als Zielstruktur nachuntersucht. Nur in beiden PCR-Verfahren negativ gebliebene Proben wurden als endgültig negativ befundet. Die Amplifikate wurden nach dem Sanger-Verfahren sequenziert. Die sequenzbasierte Erreger-Diskriminierung auf Gattungs- bis Speziesebene erfolgte in Anlehnung an die ‚Clinical Laboratory Standards Institute‘ (CLSI) Leitlinie MM18-A [17].
Ergebnisse und Diskussion
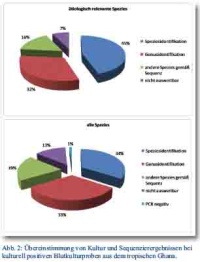
Der direkte Vergleich zwischen Kultur und molekularer Diagnostik führte zu folgendem Resultat: Von den 98 kulturell identifizierten, potenziell ätiologisch relevanten Sepsiserregern wurden fast die Hälfte auf Spezies und mehr als drei Viertel zumindest auf Gattungsebene durch die Sequenzierung bestätigt. Dabei ist eine Identifizierung auf Gattungsebene für eine kalkulierte Anpassung der Antibiotikatherapie häufig ausreichend. In weiteren mehr als 15 % der Fälle konnten inkorrekte biochemische Identifizierungsergebnisse, wie sie unter strukturschwachen tropischen Bedingungen gelegentlich vorkommen, mittels Sequenzierung korrigiert werden. In weniger als 10 % der Fälle waren die Sequenzierergebnisse so schlecht, dass keine Identifizierung gelang (Abb. 2).
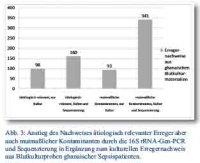
Umgekehrt konnten durch den ergänzenden Einsatz von PCR und Sequenzierung die Nachweise potenziell ätiologisch relevanter Sepsiserreger um fast zwei Drittel von 98 auf 160 Fälle erhöht werden. Allerdings stieg auch der Anteil der Kontaminationsnachweise überproportional von 93 auf 341 Proben, mutmaßlich eine Konsequenz aus eingeschränkter Sterilität während der Probenprozessierung im tropischen Routinelabor und der Kontaminationsanfälligkeit der zentrifugationsbasierten Erregeranreicherung (Abb. 3).
Die Daten machen deutlich, dass die Sepsisdiagnostik unter einfachen tropischen Bedingungen weiterer Optimierung bedarf und daher weiterhin im Fokus wehrmedizinischer Forschung bleiben sollte. Insbesondere offenbarten sich erhebliche Einschränkungen der Sensitivität der kulturellen Sepsis-Diagnostik unter ungünstigen präanalytischen Bedingungen in den Tropen. Den offenkundigen Schwächen der automatisierten Blutkulturinkubation im tropischen Umfeld wurde innerhalb des Sanitätsdienstes bereits Rechnung getragen, indem im mikrobiologischen Diagnostiklabor in Mazar-e-Sharif keine Blutkulturautomaten stehen, sondern die Blutkulturflaschen täglich visuell auf Wachstumszeichen (Trübung, Ballonierung, Bewuchs des Tauchagars etc.) inspiziert werden.
Trotz des demonstrierten erheblichen Sensitivitätszugewinns stellt auch die sequenzbasierte Erregerdiagnostik keinen Ersatz, sondern lediglich eine sinnvolle Ergänzung zur kulturellen Erregerdiagnostik dar. Resistenztestung und sich daraus ableitende Resistenzsurveillance erfordern weiterhin den kulturellen Ansatz.
Angesichts fallender Sequenzierungskosten sowie immer kleiner und handlicher werdender Sequenzierautomaten erscheint die sequenzbasierte Erregerdiagnostik im Einsatz mittelfristig in den kommenden Jahren bis Jahrzehnten als ein realisierbares Ziel. Weiterer Optimierung bedarf die kontaminationsfreie Nukleinsäureaufreinigung aus dem Blutkulturmaterial, um die Aussagekraft der diagnostischen Ergebnisse zu verbessern. Da hochsensitive panbakterielle 16S rRNA-Gen-PCR-Verfahren jedoch selbst minimale DNA-Verunreinigungen in den diagnostischen Reagenzien detektieren können [18, 19], stellt die Kontaminationsanfälligkeit eine schwer auszuschließende methodische Limitation des Verfahrens dar. Auch zur Interpretation sequenzbasierter Befunde wird im Rahmen der Sepsisdiagnostik daher mikrobiologisch-infektiologischer Sachverstand unverzichtbar bleiben.
Danksagung
Der Autor dankt den Kolleginnen und Kollegen vom Fachbereich Tropenmedizin am Bernhard-Nocht-Institut, Bundeswehrkrankenhaus Hamburg: Annett Michel, Simone Priesnitz und Ralf M. Hagen; vom Zentralen Institut der Bundeswehr in Kiel, Außenstelle Berlin, Abteilung I: Eva Halbgewachs; vom Bernhard-Nocht-Institut für Tropenmedizin in Hamburg: Denise Dekker, Jürgen May, Sven Poppert, Norbert G. Schwarz und Andreas Hahn; vom UMG-Labor der Universitätsmedizin Göttingen: Ulrike Loderstädt; vom Kumasi Center for Collaborative Research in Kumasi, Ghana: Nimako Sarpong und Kennedy Boahen; von der Kwame Nkrumah University of Science and Technology in Kumasi, Ghana: Yaw Adu-Sarkodie und vom International Vaccine Institute in Seoul, Republik Korea: Florian Marks für ihre Unterstützung der Arbeit.
Interessenkonflikt
Der Autor erklärt, dass kein Interessenkonflikt im Sinne der Richtlinien des International Committee of Medical Journal Editors besteht. Die molekulare Analytik wurde vom BMVg durch das Sonderforschungsprojekt 13K2-S-451215 ‚Neuentwicklung/Evaluierung diagnostischer molekularer Verfahren zum Nachweis von Infektionserregern und symptomorientierter Abklärung tropenmedizinisch-infektiologischer Krankheitsbilder‘ gefördert.
Literatur
- Vandersee S, Hannemann M, Herrmann F, Becker P: Multiresistente Erreger bei Patienten des deutschen Bundeswehr-Einsatzlazaretts in Masar-e Sharif. Hyg Med 2011; 36: 384-392.
- Frickmann H, Hagen RM: Bakterielle Erreger mit atypischen Resistenzmustern – ein Update. Wehrmedizinische Monatsschrift 2012; 56: 234–239.
- Helm F: Einsatzsurveillance. ISAF und die Maxime des Inspekteurs. Wehrmedizin & Wehrpharmazie 2013, 1: 40–45.
- Kumar A, Roberts D, Wood KE, et al.: Duration of hypotension before initiation of effective antimicrobial therapy is the critical determinant of survival in human septic shock. Crit Care Med 2006a; 34: 1589–1596.
- Kumar A, Safdar N, Kethireddy S, Chateau D.: A survival benefit of combination antibiotic therapy for serious infections associated with sepsis and septic shock is contingent only on the risk of death: A meta-analytic/meta-regression study. Crit Care Med 2010; 38: 1651–1664.
- Brittain-Long R, Westin J, Olofsson S, et al.: Access to a polymerase chain reaction assay method targeting 13 respiratory viruses can reduce antibiotics: a randomised, controlled trial. BMC Med 2011; 9: 44.
- Oosterheert JJ, van Loon AM, Schuurman R, et al.: Impact of rapid detection of viral and atypical bacterial pathogens by real-time polymerase chain reaction for patients with lower respiratory tract infection. Clin Infect Dis 2005; 41: 1438–1444.
- Clerc O, Prod’hom G, Vogne C, et al.: Impact of matrix-assisted laser desorption ionization time-of-flight mass spectrometry on the clinical management of patients with Gram-negative bacteremia: a prospective observational study. Clin Infect Dis 2013; 56: 1101–1107.
- Seifert H, Abele-Horn M, Fätkenheuer T, et al.: MiQ 3a Blutkulturdiagnostik. Sepsis, Endokarditis, Katheterinfektionen, Teil I. 2. Auflage Urban & Fischer München – Jena 2007; 25–27.
- Reddy EA, Shaw AV, Crump JA: Community-acquired bloodstream infections in Africa: a systemic review and meta-analysis. Lancet Infect Dis 2010; 10: 417–432.
- Deen J, von Seidlein L, Andersen F, et al.: Community-acquired bacterial bloodstream infections in developing countries in south and southeast Asia: a systematic review. Lancet Infect Dis 2012; 12: 480–487.
- Frickmann H, Dekker D, Boahen K, et al.: Increased detection of invasive enteropathogenic bacteria in pre-incubated blood culture materials by real-time PCR in comparison with automated incubation in Sub-Saharan Africa. Scan J Infect Dis 2013; 45: 616–622.
- Jenkins C, Ling CL, Ciesielczuk HL, et al.: Detection and indentification of bacteria in clinical samples by 16S rRNA gene sequencing: comparison of two different approaches in clinical practice. J Med Microbiol 2012; 61: 483–488.
- Cilia V, Lafay B, Christen R: Sequence heterogeneities among 16S ribosomal RNA sequences, and their effect on phylogenetic analyses at the species level. Mol Biol Evol 1996; 13: 451–461.
- Hagen RM, Frickmann H, Elschner M, et al.: Rapid identification of Burkholderia pseudomallei and Burkholderia mallei by fluorescence in situ hybridization (FISH) from culture and paraffin-embedded tissue samples. Int J Med Microbiol 2011; 301: 585–590.
- Rosey, AL, Abachin E, Quesnes G, et al.: Development of a broad-range 16S rDNA real-time PCR for the diagnosis of septic arthritis in children. J Microbiol Meth 2007; 68: 88–93.
- Clinical and Laboratory Standards Institute: Interpretive criteria for identification of bacteria and fungi by DNA target sequencing. Approved standard MM18-A, 1st ed. Clinical and Laboratory Standards Institute, Wayne, PA 2009; 30–34.
- Mühl H, Kochem AJ, Disqué C, Sakka SG: Activity and DNA contamination of commercial polymerase chain reaction reagents for the universal 16S rDNA real-time polymerase chain reaction detection of bacterial pathogens in blood. Diagn Microbiol Inf Dis 2010; 66: 41–49.
- Linow M: Mastermix 16S – ultra sensitive detection of microbial DNA Res Mol Microbiol 2012; 2: 1–2.
Datum: 04.12.2013
Quelle: Wehrmedizinische Monatsschrift 2013/11