SCHNELLDIFFERENZIERUNG VON KRANKHEITSERREGERN IM EINSATZ DURCH FLUORESZENZ-IN-SITU-HYBRIDISIERUNG (FISH) - CHANCEN UND PERSPEKTIVEN
Aus dem Fachbereich Tropenmedizin am Bernhard-Nocht-Institut* (Leiter: Oberfeldarzt Dr. M. Fischer) des Bundeswehrkrankenhauses Hamburg (Chefarzt: Oberstarzt Dr. J. Nakath), dem Bernhard-Nocht-Institut für Tropenmedizin, Hamburg° (Leiter: Prof. Dr. B. Fleischer, Prof. Dr. R. Horstmann und Prof. Dr. E. Tannig)
von Hagen Frickmann*, Sven Poppert° und Ralf Matthias Hagen*
Zusammenfassung
Hintergrund:
Im Einsatz kann die zeitnahe Kenntnis der Erregerspezies für eine kalkulierte Antibiotikatherapie bereits richtungsweisend sein, insbesondere wenn die lokale Resistenzsituation bekannt ist.
In der vorliegenden Arbeit wird die Fluoreszenz-In-situ-Hybridisierung (FISH) als Methode zur schnellen und einfachen Erregeridentifizierung sowie ihre mögliche Anwendung im mikrobiologischen Feldlabor beschrieben.
Methoden:
Eine selektive Literaturrecherche wurde durchgeführt. Ferner flossen die Erfahrungen der Autoren mit der Technik in die Bewertung mit ein.
Ergebnisse:
Bei der FISH erfolgt nach einer Bindung kurzer fluoreszenzmarkierter DNASonden an die Ribosomen des Erregers eine Analyse unter einem Fluoreszenzmikroskop. Dies erlaubt eine gezielte Identifizierung verschiedener Infektionserreger auf Gattungsoder Speziesebene. Eine dem Gram-Präparat nachgeschaltete FISH ist geeignet, eine umgehende Keimdifferenzierung ohne Verzögerung durch Anzucht zu gewährleisten. Derzeit ist eine breite diagnostische Anwendung der Technik im Routinelabor noch limitiert und vorerst Spezialindikationen vorbehalten. Eine zunehmende Automatisierung und Kommerzialisierung der Technik mit dem Ziel der Einführung in die Routinediagnostik ist in den kommenden Jahren zu erwarten.
Schlussfolgerungen:
Zum jetzigen Entwicklungsstand stellt die FISH-Technologie eine hilfreiche Brückentechnologie zur Schließung von Fähigkeitslücken im Einsatzlabor dar.
Rapid identifcation of pathogens in mission by fluorescence in situ hybridiziation (FISH) - chances and perspectives
Summary Introduction:
Early identification of bacteria in the field may be essential for rational antibiotic stewardship especially in case of known regional resistance patterns. The fluorescence in situ hybridization (FISH) as a method for rapid and easy identification of microbial agents and their potential use in the field laboratory will be described.
Methods:
A selective literature research was performed. The evaluation of the technique was further influenced by the experience of the authors.
Results:
Binding of short fluorescence-labeled DNA-probes to ribosomes of the infectious agents with consecutive analysis by means of fluorescence microscopy allows identification on genus or species level. FISH analysis following Gram-stain allows immediate differentiation of infectious agents without delay due to cultural growth. A broad use of the procedure in the diagnostic routine laboratory is still limited and restricted to special indications at the present time. On-going automation and commercialization of the FISH procedure are expected for the following years to guarantee compatibility with diagnostic routine.
Conclusions:
FISH has the potential to be a useful technology to close capability gaps in the field laboratory at the current stage of development.
1. Einleitung
Infektionskrankheiten zählen international bei Soldaten in Kriegs- und Krisengebieten zu den häufigsten Gründen, die zu Einschränkungen der Einsatzfähigkeit führen. Sowohl zur frühzeitigen zielgerichteten Therapieeinleitung als auch zur Früherkennung von Ausbruchsgeschehen sind Methoden zur Schnelldifferenzierung mikrobieller Erreger im Einsatz von besonderer Bedeutung. Bei bekannter lokaler Resistenzsituation beziehungsweise sorgfältigem epidemiologischem Monitoring der regionalen Resistenzlage in den Einsatzgebieten kann die zeitnahe Kenntnis der Erregerspezies für eine kalkulierte Antibiotikaempfehlung richtungsweisend sein. Dies wird aktuell durch das Auftreten multiresistenter Problemkeime, wie etwa Acinetobacter baumannii, als zusätzliche Bedrohung der ISAF-Kontingente in Afghanistan unterstrichen.
„Klassische“ Verfahren, wie zum Beispiel eine sofort nach Eingang der Proben ins Labor mögliche Gram-Färbung und lichtmikroskopische Untersuchung des Primärpräparats, erlauben bei hinreichender Keimkonzentration nur eine grobe Orientierung hinsichtlich des Erregerspektrums.
Eine endgültige mikrobiologische Erreger- Identifizierung mittels kultureller und biochemischer Verfahren sowie Resistenzbestimmungen sind zeitintensiv und nicht an allen Einsatzorten verfügbar. Die kulturelle Identifizierung setzt eine vorherige Anzucht und Isolation über 1-2 Tage voraus. Antikörperbasierte Differenzierungsmethoden (Latex- Agglutinationstests und Immunfluoreszenzverfahren) stehen nur für ausgewählte Erreger zur Verfügung und sind zudem nicht bei allen Primärmaterialien anwendbar. Aufgrund durchweg erheblicher Limitationen hinsichtlich der Sensitivität und Spezifität konnten sich diese Techniken in der Routinediagnostik nicht breit durchsetzen. Eine molekularbiologische Schnelldiagnostik mittels der Nukleinsäure-Amplifikationstechniken ist bislang im Einsatzlabor, abgesehen von der Influenza-PCR-Diagnostik 2009 (3), noch nicht verfügbar.
2. Methoden
Ein schnell und einfach durchzuführendes molekularbiologisches Verfahren zur Identifizierung von Erregern (Bakterien, Pilzen, Protozoen) ist die Fluoreszenz-In-situ-Hybridisierung (FISH). Anhand einer selektiven Literaturrecherche unter Einbeziehung der Datenbank „pubmed“ (www.pubmed.com) wurde untersucht, ob sich die FISH zur schnellen Identifizierung von Krankheitserregern im Feldlabor eignet. Die anhand selbst durchgeführter Validationsstudien (2, 7, 10) erworbene Erfahrung der Autoren mit der FISH floss in die Bewertung der Technik ein.
3. Ergebnisse und Diskussion
3.1 Funktionsprinzip und Durchführung der FISH
Bei der FISH erfolgt die Gattungs- oder Speziesbestimmung durch die Bindung kurzer fluoreszenzmarkierter erregerspezifischer DNA-Sonden an die Ribosomen des Erregers mit anschließender Analyse unter dem Fluoreszenzmikroskop. Nach Fixierung auf konventionellen Objektträgern und Probenvorbehandlung, zum Beispiel vorgeschalteter Permeabilisierung bei Gram-positiven Erregern, erfolgt die Hybridisierung. Dabei lagert sich die Sonde an die komplementäre ribosomale RNA an. Überschüssige Sonden werden anschließend in einem Waschschritt entfernt. Neben der spezifischen Sonde kommt immer auch eine unspezifische Eubakteriensonde zur Anwendung, die mit einem anderen Fluoreszenzfarbstoff markiert ist.
Bei Sprosspilzverdacht wird eine sogenannte Hefenkonsensussonde eingesetzt. Eubakterien- oder Hefenkonsensussonde dienen als interne Kontrollen, um den fehlerfreien Verlauf der jeweiligen Färbung zu demonstrieren (1, 2). Mit DAPI (4’,6-Diamidino- 2-phenylindol) wird zusätzlich die gesamte DNA gefärbt. Unspezifische Bindungen der Sonden an DNAfreien Detritus sind so zu erkennen. Die Detektion der Fluoreszenzsignale der spezifischen und unspezifischen Sonde sowie der DAPI-Färbung erfolgt über verschiedene Farbkanäle des Fluoreszenzmikroskops. Für das genaue technische Prozedere sei auf eine vorausgegangene Arbeit verwiesen (2).
Die einzelnen Arbeitsschritte der FISH und Anwendungsbeispiele sind in den Abbildungen 1 und 2 dargestellt.
Abb 1: Technische Durchführung der FISH. Graue Ellipsen: Bakterien, braune Doppelellipsen: Ribosomen, gelbe Striche mit roten Punkten: markierte Oligonukleotide. (Fotos und Design: modifiziert nach Prof. Dr. Matthias Horn, Universität Wien)
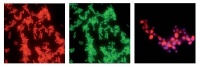
Abb 2 a: Bindung der gattungsspezifischen Cy3-markierten Acinetobacter spp.-Sonde an A. baumannii (rote Fluoreszenz). 2 b: Bindung der FAM-markierten Eubakterien-Sonde an A. baumannii im Sinne einer internen Kontrolle (grüne Fluoreszenz). 2 c: Bindung einer Cy3-markierten Candida albicans spezifischen Sonde (rote Fluoreszenz) an C. albicans sowie Bindung des nicht interkalierenden DNA-Farbstoffs DAPI (blaue Fluoreszenz) an die Pilz-DNA zum Ausschluss unspezifischer Bindung an nicht-DNA-haltigen Detritus (hier digitale Überlagerung beider Farbkanäle). (Fotos: Dr. H. Frickmann, Dr. S. Poppert und Dr. R. M. Hagen)
3.2 Vorteile der Methode und daraus resultierende Einsatzmöglichkeiten
Zu den offenkundigen Vorteilen der FISH gehört die schnelle (< 60 Minuten) Durchführbarkeit. Verfahren zur FISH-basierten Schnelldifferenzierung wurden bereits für Erreger lebensbedrohlicher Erkrankungen, wie Pseudomonaden, Stenotrophomonas maltophilia, Enterobakterien, Meningokokken, Pneumokokken und Staphylococcus aureus aus Blutkulturen und anderen wichtigen klinischen Materialien beschrieben (2). Insbesondere die zeitnahe Erregerdifferenzierung aus Blutkulturen kann die Entscheidung zur Umstellung der kalkulierten antibiotischen Therapie bei Sepsis-Patienten beeinflussen und damit potenziell Menschenleben retten.
Eine In-situ-FISH zum Erregerdirektnachweis im Primärpräparat ist mit wenigen Modifikationen realisierbar (2). Vergleichbar dem Gram-Präparat ist dabei auch eine Semiquantifizierung möglich. Die FISH erfordert wenig technisches Equipment und ist somit prinzipiell auch im Feldlabor anwendbar. Die Materialkosten liegen – beim Einsatz publizierter, patentfreier DNASonden – bei unter 1 € pro Reaktion. Selbstverständlich ermöglicht die FISH auch die umgehende Erregerdifferenzierung von Kulturmaterial, wenn eine biochemische Identifizierung in zeitkritischen Fällen nicht abgewartet werden kann oder die Erreger phänotypisch schwierig zu differenzieren sind (2, 4, 5, 7, 9). Diese molekularbiologische Methode wird nicht durch Probleme, wie schwierige Anzucht beziehungsweise zweifelhafte Substratumsetzung, limitiert, die bei der biochemischen Differenzierung auftreten können.
Als mittelfristige Indikationen für die Anwendung der FISH betrachten wir Primärpräparate von primär sterilen Materialien, zum Beispiel Liquor und Punktate, oder von klinisch hoch relevanten Materialien, zum Beispiel Proben von chronischen Wunden und invasiv gewonnene Proben aus den tiefen Atemwegen (Trachealsekret und BAL) sowie schwer vereinzelbare Mischkulturen. In der aktuellen Literatur finden sich auch Hinweise auf die Möglichkeit der Detektion von biofilmbildendem Pseudomonas aeruginosa in Gewebe von kulturnegativen chronischen Wunden (6), sodass die FISH hinsichtlich ihrer Sensitivität in diesem Spezialfall selbst der Kultur überlegen ist.
3.3 Sensitivität, Modus der Anwendung sowie Zeitbedarf der Methode
Die Sensitivität (Nachweisgrenze) der FISH im Primärpräparat liegt nach unserer Erfahrung etwa im Bereich der Gram-Färbung (Grenze bei 100 000 Kolonie-bildenden-Einheiten/ml). Dies lässt den diagnostischen Einsatz der FISH bei vorher negativem Gram-Präparat nicht erfolgversprechend erscheinen. Ein vorgeschaltetes Gram-Präparat ist auch erforderlich, um Sondenauswahl und Permeabilisierungsbedingungen festzulegen.
Der diagnostische Einsatz der FISH erfordert einen strukturierten Algorithmus. Das heißt, es muss vorab klar sein, nach welchen Erregern gesucht werden soll. Das Aufbringen der zu untersuchenden Materialien erfolgt auf Mehrfeldobjektträger, was die zeitgleiche Anwendung verschiedener Sonden ermöglicht. Dieses sogenannte „multiple probe concept“ setzt jedoch voraus, dass die entsprechenden Sonden unter gleichen Reaktionsbedingungen validiert wurden.
Als Zeitansatz sollten bei Sprosspilzen und Gram-negativen Erregern eine Stunde, bei Gram-positiven Erregern 1,5 Stunden kalkuliert werden, wobei das technische Personal aufgrund kurzer Inkubationszeiten mit repetitivem Interventionsbedarf auch durchgehend gebunden ist. Die notwendige Ausstattung (Abb 1) ist überschaubar und lässt sich in einem Laborcontainer unterbringen.
3.4 Spezifität und Limitierungen der Methode
Vor Einführung in die diagnostische Routine sind umfassende Validationsstudien angezeigt, da viele in der Literatur beschriebene Forschungssonden für wissenschaftliche Fragestellungen zwar hinreichend sensitiv, für die Routinediagnostik aber nicht spezifisch genug sind (2, eigene Erfahrungen der Autoren). Bei der FISH gibt es nur ein Nukleinsäure-bindendes Element, während bei einer real-time-PCR durch 2 oder 3 Oligonukleotide eine ungleich höhere Spezifität gewährleistet ist. Zu erwartende Kreuzreaktionen, wie sie typischerweise bei Fehlpaarungen von 2 Basenpaaren auftreten, müssen dem Untersucher bekannt sein, um FISH-Befunde interpretieren zu können.
Die Aktualität und Spezifität der Sonden sollte daher auch MIQ-gerecht regelmäßig, beispielsweise mittels der Software „probecheck“ (www.microbial- ecology.net/probecheck/), überprüft werden. Auch hier kann das „multiple probe concept“ dazu beitragen, durch den kombinierten Einsatz mehrerer Sonden Unspezifitäten einzelner Oligonukleotidbindungen zu erkennen. Nachteile der Methode liegen in der Abhängigkeit von einem erfahrenen Untersucher und in der Subjektivität der Auswertung, da das Verfahren (noch) nicht standardisiert ist. Ferner liegt die Sensitivität bei Analysen vom Primärpräparat unter der von PCR-Verfahren. Auch erfordert die FISH aufgrund der Spezifität der Sonden eine zielgerichtete Suche, was – bei beschränktem Sondensatz – im Einzelfall nur eine Ausschlussdiagnostik ermöglicht. Dies stellt einen Nachteil im Vergleich zur nichtselektiv durchführbaren kulturellen Anzucht dar.
Die exzellenten Sensitivitäten und Spezifitäten, wie sie für die FISH vom klinischen Material in der Literatur beschrieben sind (4, 5, 7, 9, 10), setzen, wie bei jeder anderen Labormethode auch, eine technisch einwandfreie Durchführung durch gut eingearbeitetes Personal voraus. Zu beachten sind bei dem grundsätzlich robusten Verfahren unter anderem die Lichtempfindlichkeit der Sonden, die Flüchtigkeit von Pufferbestandteilen (insbesondere des Formamids) sowie die Temperaturanfälligkeit der Reaktionen. Dies erfordert ein zügiges Arbeiten. Darüber hinaus bergen multiple Einzelschritte das Risiko von Pipettierfehlern in sich. Während der Waschschritte kann es zur partiellen Ablösung des fixierten Materials vom Objektträger mit entsprechendem Sensitivitätsverlust kommen. Bei Mehrfeldobjektträgern besteht dabei die Gefahr von Kreuzkontaminationen durch das „Abschwimmen“ von Material von einem Feld aufs nächste. Die Verwendung toxischer Reagenzien, namentlich Formamid, DAPI und SDS (Natriumdodecylsulfat), macht einen sorgfältigen Umgang unter Beachtung des Arbeitsschutzes erforderlich.
4. Schlussfolgerungen
Aktueller Stand der Anwendung in der Bundeswehr
Das Einsatzspektrum der Bundeswehr in Regionen mit komplexem Erregerspektrum sowie die lokalen Gegebenheiten mit oftmals limitierten Ressourcen erfordern schnelles Handeln. Im Hinblick auf die labormedizinisch-mikrobiologische Einsatzbereitschaft bestehen weiterhin Fähigkeitslücken, die lediglich durch Einführung und Anwendung moderner Methoden kompensierbar sind. Weder der Betrieb eines vollständigen mikrobiologischen Feldlabors an allen Einsatzorten noch eine kurzfristige Abstützung auf Nukleinsäure- Amplifikationstechniken in der Infektionsdiagnostik erscheinen derzeit umsetzbar.
Auch mit FISH-basierter Erregerdiagnostik gibt es bislang noch wenig Erfahrung in mikrobiologischen Laboratorien der Bundeswehr. Es existieren spezifische Sonden zum Nachweis von BSchutz- relevanten Erregern, wie Brucellen (10) und Francisellen (8). Darüber hinaus konnte die FISH-Technik am Fachbereich Tropenmedizin des Bernhard- Nocht-Instituts (Bundeswehrkrankenhaus Hamburg) etabliert werden. Zu den Aufgaben der Laborgruppe des Fachbereichs gehört es, verfügbare moderne Techniken zu etablieren, zu evaluieren, gegebenenfalls um neue Anwendungsfelder zu ergänzen und für Einsätze in den Tropen nutzbar zu machen. Dies geschieht in der Regel in zivil-militärischer Zusammenarbeit auf der Basis des bestehenden Kooperationsvertrags mit Partnern am Bernhard- Nocht-Institut. Im Stadium der Validation an klinischem Material befinden sich dort entwickelte Sonden zum Nachweis schwierig zu identifizierender Erreger, beispielsweise Haemophilus spp. und Candida spp., der potenziell multiresistenten Acinetobacter spp. sowie einsatzrelevanter Erreger von Tropenkrankheiten wie Leishmania spp. und Burkholderia pseudomallei.
Der entscheidende Vorteil der FISH besteht in der zeitnahen Erregerdifferenzierung unmittelbar im Direktpräparat in weniger als 2 Stunden im Vergleich zur biochemischen Differenzierung nach Anzucht über mehrere Tage (!). Daher sehen wir in der FISH eine hilfreiche Brückentechnik zum kurzfristigen Schließen von Fähigkeitslücken bis andere Nachweisverfahren, wie zum Beispiel die bereits erwähnten PCR-Verfahren, auf breiter Basis im Einsatz verfügbar sind (3). In diesem Kontext stellt die dem Gram-Präparat nachgeschaltete FISH (2, 4, 7, 9) als kostengünstige und einfach durchzuführende Methode zur orientierenden Schnelldiagnostik eine nützliche Ergänzung zum vorhandenen Methodenspektrum für das mikrobiologische Feldlabor dar.
Auch andere moderne Entwicklungen wie die MALDI-TOF-MS-Analyse (Matrix Assisted Laser Desorption/Ionisation– Time Of Flight-Massenspektrometrie) oder auch die Nukleinsäure-Sequenzierung, zum Beispiel Pyrosequenzierung, könnten in den nächsten Jahren oder Jahrzehnten als feldtaugliche Methoden im Einsatz zur Verfügung stehen. Ihre weitere Entwicklung bleibt abzuwarten. Durch eine zunehmende Automatisierung und Kommerzialisierung der FISH-Technik durch Firmen, die bereits heute breite diagnostische FISHPanels anbieten, wird das Verfahren vermutlich zunehmend Einzug in die mikrobiologische Routinediagnostik erfahren. Breit angelegte Validationsstudien dieser Systeme stehen bisher aus, sind jedoch für die kommenden Jahre zu erwarten.
Bis dahin muss auf intern validierte Verfahren zurückgegriffen werden. Wie bei anderen fluoreszenzbasierten Färbetechniken ist die Bewertung von FISH-Resultaten am Primärparat subjektiv und untersucherabhängig. Aufgrund unspezifischer Bindungen an Probenmaterial wird eine automatisierte Auswertung erschwert bis unmöglich gemacht. Somit erfordert die Beurteilung von FISH-Ergebnissen neben technischer Standardisierung hohe fachliche Expertise. Diese kann über die Telemikrobiologie für die Auswertung der FISH durch entsprechend qualifiziertes und in der Technik erfahrenes Personal auch für die Einsatzlabore grundsätzlich gewährleistet werden.
Die FISH ist eine einfach durchzuführende, kostengünstige Methode zur Schnelldifferenzierung mikrobieller Erreger im Einsatz, die bei bekannter lokaler Resistenzsituation gegebenenfalls die Entscheidung für eine gezielte Anpassung der kalkulierten Antibiotikatherapie beeinflusst. Eine Standardisierung und Kommerzialisierung der Technik zur mittelfristigen Verwendung im Feldlabor erscheint sinnvoll und indiziert.
Literatur:
- Amann RI, Krumholz L, Stahl DA: Fluorescent-oligonucleotide probing of whole cells for determinative, phylogenetic, and environmental studies in microbiology. J Bacteriol 1990; 172:762-770.
- Frickmann H, Crusius S, Podbielski A: Fluoreszenz-in-situ-Hybridisierung (FISH) vom mikroskopischen Primärpräparat – Vor- und Nachteile sowie technische Hinweise. Mikrobiologe 2010; 20:95-101.
- Hagen RM, Wiemer D, Priesnitz S, Fischer M: Perspektiven moderner PCR-Diagnostik in der Tropenmedizin. Wehrmedizin und Wehrpharmazie 2010; 3:45-48.
- Hogardt M, Trebesius K, Geiger AM, Hornef M, Rosenecker J, Heesemann J: Specific and rapid detection by fluorescent in situ hybridization of bacteria in clinical samples obtained from cystic fibrosis patients. J Clinical Microbiol 2000; 38:818-825.
- Kempf VAJ, Trebesius K, Autenrieth IB: Fluorescent in situ hybridisation allows rapid identification of microorganisms in blood cultures. J Clin Microbiol 2000; 38:830-838.
- Kirketerp-Møller K, Jensen PØ, Fazli M et al.: Distribution, organization, and ecology of bacteria in chronic wounds. J Clin Microbiol 2008; 46:2717-2722.
- Poppert S, Essig A, Stoehr B et al.: Rapid diagnosis of bacterial meningitis by real-time PCR and fluorescence in situ hybridization. J Clin Microbiol 2005; 43:3390-3397.
- Splettstoesser WD, Seibold E, Zeman E, Trebesius K, Podbielski A: Rapid differentiation of Francisella species and subspecies by fluorescent in situ hybridization targeting the 23S rRNA. BMC Microbiology 2010; 10:72.
- Trebesius K, Leitritz L, Adler K, Schubert S, Autenrieth IB, Heesemann J: Culture independent and rapid identification of bacterial pathogens in necrotizing fasciitis and streptococcal toxic shock syndrome by fluorescence in situ hybridization. Med Microbiol Immunol 2000; 188:169-175.
- Wellinghausen N, Nöckler K, Sigge A, Bartel M, Essig A, Poppert S: Rapid detection of Brucella spp. in blood cultures by fluorescence in situ hybridization. J Clin Microbiol 2006; 44:1828-1830.
Datum: 25.06.2011
Quelle: Wehrmedizinische Monatsschrift 2011/4